เป็นที่ทราบกันดีว่าในความเชื่อหลัก RNA เป็นตัวกลางในการถอดรหัสระหว่างการแสดงออกของ DNA และโปรตีนเมื่อเปรียบเทียบกับการตรวจจับ DNA การตรวจจับ RNA สามารถสะท้อนการแสดงออกของยีนในสิ่งมีชีวิตได้อย่างเป็นกลางมากกว่าการทดลองที่เกี่ยวข้องกับ RNA ได้แก่: qRT-PCR, RNA-Seq และการตรวจจับยีนฟิวชัน เป็นต้น จากลักษณะเฉพาะของ RNA เอง (วงแหวนน้ำตาลของ RNA มีหมู่ไฮดรอกซิลอิสระมากกว่าวงแหวนน้ำตาลของ DNA) ประกอบกับ RNases จำนวนมากในสิ่งแวดล้อม RNA จึงไม่เสถียรและย่อยสลายได้ง่ายกว่า DNAขยะเข้าขยะออก ถ้าคุณภาพของ RNA ไม่ดี ผลการทดลองจะต้องไม่น่าพอใจ โดยเฉพาะอย่างยิ่งแสดงว่าเป็นข้อมูลที่ไม่ถูกต้องหรือความสามารถในการทำซ้ำได้ไม่ดีดังนั้นควรให้ความสนใจกับการประมวลผลของ RNA มากขึ้น และการเชื่อมโยงของการควบคุมคุณภาพก็มีความสำคัญมากขึ้นเช่นกัน เพื่อให้มั่นใจถึงความแม่นยำและความถูกต้องของข้อมูลการทดลองที่ตามมา
สำหรับการควบคุมคุณภาพของ RNA โดยทั่วไปมีวิธีที่ใช้กันโดยทั่วไปดังต่อไปนี้:
- สเปกโตรโฟโตเมตรี
- agarose gel อิเล็กโทรโฟรีซิส
- เครื่องวิเคราะห์ชีวภาพแบบอจิเลนต์
- PCR เชิงปริมาณฟลูออเรสเซนต์แบบเรียลไทม์
- วิธีย้อมเรืองแสง Qubit
01 สเปกโตรโฟโตเมตรี
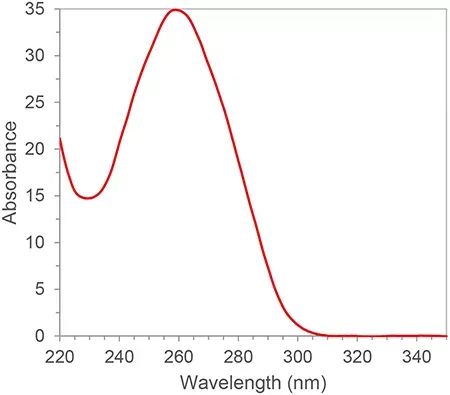
RNA มีการเชื่อมพันธะคู่เข้าด้วยกันและมีจุดสูงสุดในการดูดกลืนแสงที่ความยาวคลื่น 260 นาโนเมตรตามกฎของแลมเบิร์ต-เบียร์ เราสามารถคำนวณความเข้มข้นของ RNA จากจุดสูงสุดในการดูดซับที่ 260 นาโนเมตรนอกจากนี้ เรายังสามารถคำนวณความบริสุทธิ์ของ RNA ตามอัตราส่วนของพีคการดูดซับ 260nm, 280nm และ 230nm280 นาโนเมตรและ 230 นาโนเมตรเป็นจุดสูงสุดของการดูดซึมของโปรตีนและโมเลกุลขนาดเล็กตามลำดับอัตราส่วนของ A260/A280 และ A260/A230 ของความบริสุทธิ์ของ RNA ที่ผ่านการรับรองควรมากกว่า 2 ถ้าน้อยกว่า 2 แสดงว่ามีโปรตีนหรือโมเลกุลขนาดเล็กปนเปื้อนในตัวอย่าง RNA และต้องทำให้บริสุทธิ์อีกครั้งแหล่งที่มาของการปนเปื้อนจะส่งผลต่อการทดลองขั้นปลาย เช่น การยับยั้งประสิทธิภาพการขยายของปฏิกิริยา PCR ทำให้ได้ผลลัพธ์เชิงปริมาณที่ไม่ถูกต้องความบริสุทธิ์ของ RNA มีอิทธิพลอย่างมากต่อผลลัพธ์ที่ตามมา ดังนั้น สเปกโตรโฟโตเมตรีโดยทั่วไปจึงเป็นตัวควบคุมคุณภาพที่ขาดไม่ได้ในขั้นตอนแรกของการทดลองกรดนิวคลีอิก
รูปที่ 1 สเปกตรัมการดูดซึม RNA/DNA ทั่วไป
02 อะกาโรสเจล อิเล็กโตรโฟรีซิส
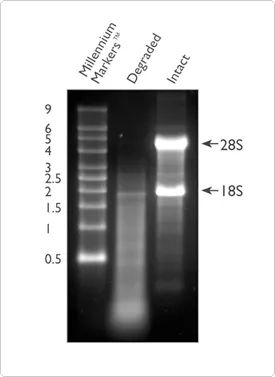
นอกจากความบริสุทธิ์แล้ว ความสมบูรณ์ของ RNA ยังเป็นหนึ่งในตัวบ่งชี้ที่สำคัญสำหรับการตัดสินคุณภาพของ RNAการสลายตัวของ RNA จะนำไปสู่ชิ้นส่วนสั้นจำนวนมากในตัวอย่าง ดังนั้นจำนวนชิ้นส่วน RNA ที่สามารถตรวจจับได้อย่างมีประสิทธิภาพและครอบคลุมโดยลำดับการอ้างอิงจะลดลงสามารถตรวจสอบความสมบูรณ์ของ RNA ได้โดยอิเล็กโตรโฟรีซิสของ RNA ทั้งหมดบนเจลอะกาโรส 1%วิธีนี้สามารถกำหนดค่าเจลด้วยตัวคุณเอง หรือใช้ระบบ E-Gel™ สำเร็จรูปสำหรับการทดสอบความสมบูรณ์มากกว่า 80% ของ RNA ทั้งหมดคือ RNA ของไรโบโซม ซึ่งส่วนใหญ่ประกอบด้วย 28S และ 18S rRNA (ในระบบของสัตว์เลี้ยงลูกด้วยนม)RNA ที่มีคุณภาพดีจะแสดงแถบสว่างที่ชัดเจนสองแถบ ซึ่งเป็นแถบสว่าง 28S และ 18S ตามลำดับ ที่ 5 Kb และ 2 Kb และอัตราส่วนจะมีแนวโน้มใกล้เคียงกับ 2:1หากอยู่ในสถานะกระจาย แสดงว่าตัวอย่าง RNA อาจถูกย่อยสลาย และขอแนะนำให้ใช้วิธีการที่อธิบายในภายหลังเพื่อทดสอบคุณภาพของ RNA เพิ่มเติม
รูปที่ 2 การเปรียบเทียบของ RNA ที่เสื่อมสภาพ (เลน 2) และ RNA ที่ไม่เสียหาย (เลน 3) บน agarose gel electrophoresis
03 เครื่องวิเคราะห์ชีวภาพ Agilent
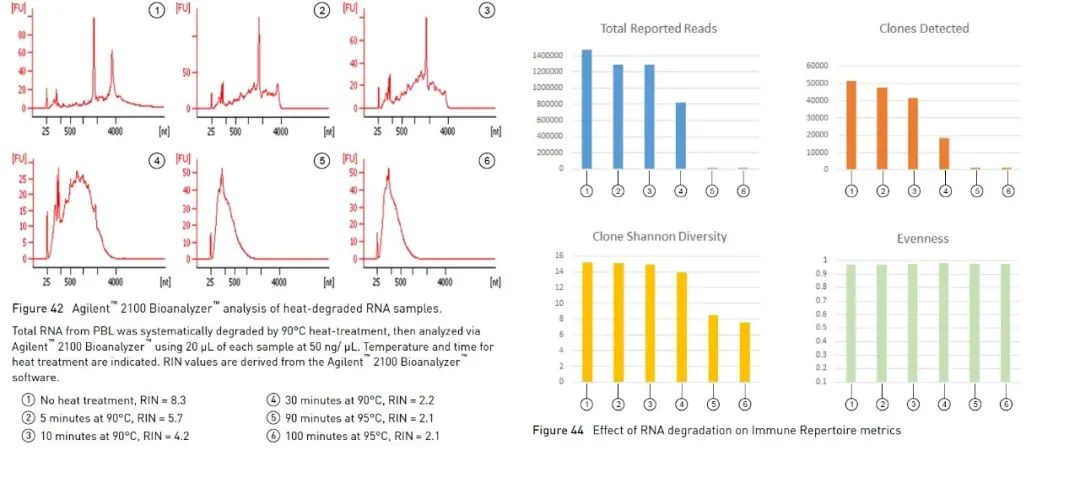
นอกจากวิธี agarose gel electrophoresis ที่อธิบายไว้ข้างต้นแล้ว ซึ่งสามารถช่วยให้เราระบุความสมบูรณ์ของ RNA ได้ง่ายและรวดเร็ว เรายังสามารถใช้ Agilent bioanalyzer เพื่อระบุความสมบูรณ์ของ RNA ได้อีกด้วยใช้การผสมผสานระหว่างไมโครฟลูอิดิกส์ อิเล็กโตรโฟรีซิสของเส้นเลือดฝอย และการเรืองแสงเพื่อประเมินความเข้มข้นและความสมบูรณ์ของอาร์เอ็นเอด้วยการใช้อัลกอริทึมในตัวเพื่อวิเคราะห์โปรไฟล์ของตัวอย่าง RNA เครื่องวิเคราะห์ทางชีวภาพ Agilent สามารถคำนวณค่าความสมบูรณ์ของ RNA อ้างอิง หมายเลขความสมบูรณ์ของ RNA (ต่อไปนี้จะเรียกว่า RIN) [1]ยิ่งค่าของ RIN สูง ความสมบูรณ์ของ RNA ก็จะยิ่งสูงขึ้น (1 คือเสื่อมโทรมมาก 10 คือสมบูรณ์ที่สุด)การทดลองบางอย่างเกี่ยวกับ RNA แนะนำให้ใช้ RIN เป็นพารามิเตอร์สำหรับการประเมินคุณภาพยกตัวอย่างการทดลองหาลำดับปริมาณงานสูง (ต่อไปนี้เรียกว่า NGS) แนวทางของ Oncomine™ Human Immune Repertoire ซึ่งใช้ในการตรวจหาตัวรับบีเซลล์และทีเซลล์แอนติเจนในชุดแผง Oncomine ของ Thermo Fisher แนะนำว่าตัวอย่างที่มีค่า RIN มากกว่า 4 สามารถวัดค่าการอ่านและโคลนที่มีประสิทธิภาพมากขึ้น (รูปที่ 3)มีช่วงที่แนะนำที่แตกต่างกันสำหรับพาเนลต่างๆ และบ่อยครั้ง RIN ที่สูงขึ้นจะทำให้ข้อมูลมีประสิทธิภาพมากขึ้น
รูปที่ 3 ในการทดลอง Oncomine™ Human Immune Repertoire ตัวอย่างที่มีค่า RIN มากกว่า 4 สามารถตรวจจับการอ่านที่มีประสิทธิภาพมากขึ้นและการโคลน T เซลล์【2】
อย่างไรก็ตาม ค่า RIN ก็มีข้อจำกัดเช่นกันแม้ว่า RIN จะมีความสัมพันธ์สูงกับคุณภาพของข้อมูลการทดลอง NGS แต่ก็ไม่เหมาะสำหรับตัวอย่าง FFPEตัวอย่าง FFPE ได้รับการบำบัดทางเคมีเป็นเวลานาน และโดยทั่วไปแล้ว RNA ที่สกัดออกมามีค่า RIN ค่อนข้างต่ำอย่างไรก็ตาม ไม่ได้หมายความว่าข้อมูลที่มีประสิทธิภาพของการทดสอบจะต้องไม่เป็นที่น่าพอใจในการประเมินคุณภาพของตัวอย่าง FFPE อย่างแม่นยำ เราจำเป็นต้องใช้การวัดอื่นที่ไม่ใช่ RINนอกจาก RIN แล้ว เครื่องวิเคราะห์ชีวภาพ Agilent ยังสามารถคำนวณค่า DV200 เป็นพารามิเตอร์การประเมินคุณภาพ RNA ได้อีกด้วยDV200 เป็นพารามิเตอร์ที่คำนวณสัดส่วนของชิ้นส่วนที่มีขนาดใหญ่กว่า 200 bp ในตัวอย่าง RNADV200 เป็นตัวบ่งชี้คุณภาพตัวอย่าง FFPE ที่ดีกว่า RINสำหรับ RNA ที่สกัดโดย FFPE นั้นมีความสัมพันธ์สูงมากกับจำนวนยีนที่สามารถตรวจจับได้อย่างมีประสิทธิภาพและความหลากหลายของยีน [3]แม้ว่า DV200 สามารถชดเชยข้อบกพร่องในการตรวจจับคุณภาพของ FFPE ได้ แต่เครื่องวิเคราะห์ทางชีวภาพ Agilent ก็ยังไม่สามารถวิเคราะห์ปัญหาคุณภาพในตัวอย่าง RNA ได้อย่างครอบคลุม รวมถึงดูว่ามีสารยับยั้งในตัวอย่างหรือไม่ตัวยับยั้งเองอาจส่งผลต่อประสิทธิภาพการขยายของการทดลองขั้นปลาย และลดปริมาณข้อมูลที่เป็นประโยชน์เพื่อให้ทราบว่ามีสารยับยั้งในตัวอย่างหรือไม่ เราสามารถใช้วิธี PCR เชิงปริมาณเรืองแสงตามเวลาจริงซึ่งจะอธิบายต่อไป
04 PCR เชิงปริมาณฟลูออเรสเซนต์แบบเรียลไทม์
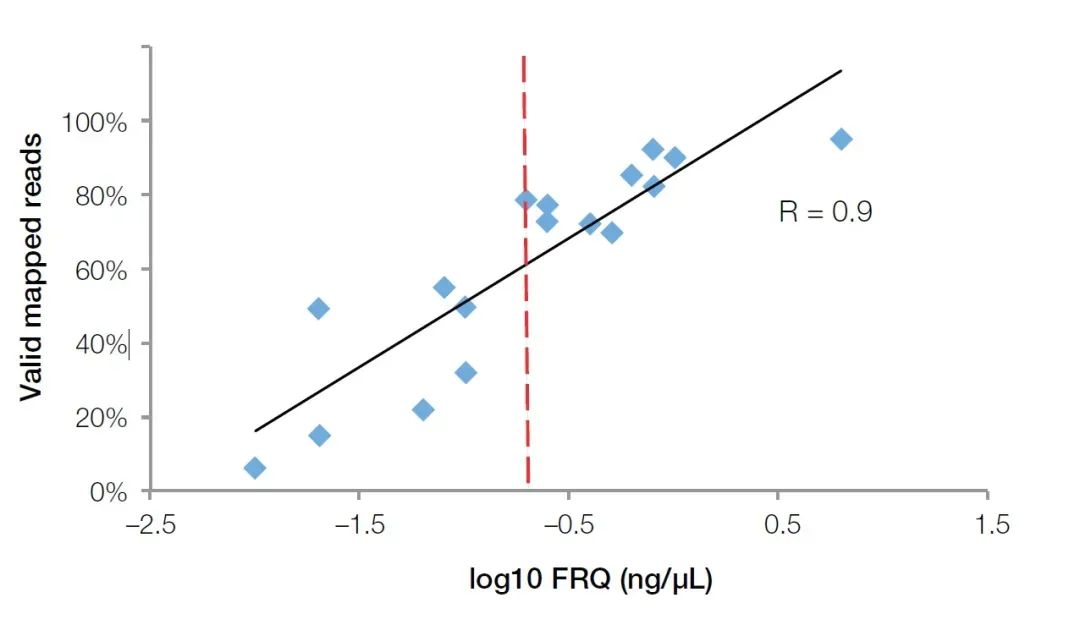
วิธี PCR เชิงปริมาณเรืองแสงตามเวลาจริงไม่เพียงแต่ตรวจจับสารยับยั้งในตัวอย่างเท่านั้น แต่ยังสะท้อนถึงคุณภาพของ RNA ในตัวอย่าง FFPE ได้อย่างแม่นยำอีกด้วยเมื่อเปรียบเทียบกับเครื่องวิเคราะห์ทางชีวภาพ Agilent เครื่องมือเชิงปริมาณฟลูออเรสเซนซ์ตามเวลาจริงจะได้รับความนิยมมากกว่าในห้องปฏิบัติการทางชีววิทยาที่สำคัญ เนื่องจากการใช้งานที่กว้างขวางกว่าในการทดสอบคุณภาพของตัวอย่าง RNA เราจำเป็นต้องซื้อหรือเตรียมไพรเมอร์โพรบสำหรับยีนอ้างอิงภายในเท่านั้น เช่น GUSB (Cat no. Hs00939627)ด้วยการใช้ไพรเมอร์ โพรบ และมาตรฐาน (RNA ทั้งหมดของความเข้มข้นที่ทราบ) เพื่อทำการทดลองเชิงปริมาณสัมบูรณ์ ความเข้มข้นของชิ้นส่วน RNA ที่มีประสิทธิภาพสามารถคำนวณเป็นมาตรฐานการประเมินคุณภาพ RNA (Functional RNA Quantitation (FRQ) หรือเรียกสั้นๆ ว่า FRQ)ในการทดสอบ NGS เราพบว่า FRQ ของตัวอย่าง RNA มีความสัมพันธ์สูงมากกับปริมาณข้อมูลที่มีประสิทธิภาพสำหรับตัวอย่างทั้งหมดที่มากกว่า 0.2ng/uL FRQ อย่างน้อย 70% ของการอ่านสามารถครอบคลุมลำดับอ้างอิงได้อย่างมีประสิทธิภาพ (รูปที่ 4)
รูปที่ 4 ค่า FRQ ที่ตรวจพบโดยวิธีการเชิงปริมาณของฟลูออเรสเซนซ์มีความสัมพันธ์ที่สูงมาก (R2>0.9) กับข้อมูลที่มีประสิทธิภาพที่ได้รับจากการทดลอง NGSเส้นสีแดงคือค่า FRQ เท่ากับ 0.2 ng/uL (log10 = -0.7)【4】
นอกจากจะใช้ได้กับตัวอย่าง FFPE แล้ว วิธี PCR เชิงปริมาณแบบเรียลไทม์ยังสามารถตรวจสอบสารยับยั้งในตัวอย่างได้อย่างมีประสิทธิภาพเราสามารถเพิ่มตัวอย่างที่จะตรวจจับเข้าไปในระบบปฏิกิริยาด้วย Internal Positive Control (IPC) และ Assay จากนั้นทำการหาปริมาณฟลูออเรสเซนซ์เพื่อให้ได้ค่า Ctถ้าค่า Ct ช้ากว่าค่า Ct ในปฏิกิริยาที่ไม่มีตัวอย่าง แสดงว่ามีสารยับยั้งอยู่ในตัวอย่างและยับยั้งประสิทธิภาพการขยายในปฏิกิริยา
05 วิธีย้อมเรืองแสง Qubit
Qubit Fluorometer เป็นอุปกรณ์ขนาดเล็กที่ใช้บ่อยที่สุดสำหรับการตรวจจับความเข้มข้นของกรดนิวคลีอิกและความบริสุทธิ์ ซึ่งใช้งานง่ายและมีอยู่ในห้องปฏิบัติการอณูชีววิทยาเกือบทุกแห่งเครื่องจะคำนวณความเข้มข้นของกรดนิวคลีอิกอย่างแม่นยำโดยการตรวจจับและสีย้อมเรืองแสงที่จับกับกรดนิวคลีอิก (สารทำปฏิกิริยาการตรวจจับ Qubit)Qubit มีความไวและความจำเพาะสูง และสามารถหาปริมาณ RNA ได้อย่างแม่นยำจนถึงความเข้มข้น pg/µLนอกจากความสามารถที่รู้จักกันดีในการหาปริมาณความเข้มข้นของกรดนิวคลีอิกอย่างแม่นยำแล้ว Qubit 4.0 รุ่นใหม่ล่าสุดของ Thermo Fisher ยังสามารถตรวจจับความสมบูรณ์ของ RNA ได้อีกด้วยระบบตรวจจับ RNA ของ Qubit 4.0 (RNA IQ Assay) ตรวจจับความสมบูรณ์ของ RNA โดยตรวจจับสีเรืองแสงเฉพาะสองสีพร้อมกันสีย้อมเรืองแสงทั้งสองนี้สามารถจับกับชิ้นส่วนขนาดใหญ่และชิ้นส่วนขนาดเล็กของ RNA ตามลำดับสีย้อมเรืองแสงทั้งสองนี้ระบุสัดส่วนของชิ้นส่วนขนาดใหญ่ของ RNA ในตัวอย่าง และจากค่านี้ จะสามารถคำนวณค่า IQ (ความสมบูรณ์และคุณภาพ) ที่แสดงถึงคุณภาพของ RNA ได้ค่า IQ ใช้ได้กับทั้งตัวอย่าง FFPE และไม่ใช่ FFPE และมีอิทธิพลอย่างมากต่อคุณภาพการจัดลำดับที่ตามมายกตัวอย่างการทดลอง NGS ในการทดสอบ RNA-Seq ที่ดำเนินการบนแพลตฟอร์ม Ion torrent™ ตัวอย่างส่วนใหญ่ที่มีค่า IQ มากกว่า 4 มีการอ่านที่มีประสิทธิภาพอย่างน้อย 50% (รูปที่ 5)เมื่อเปรียบเทียบกับวิธีการตรวจจับที่กล่าวถึงข้างต้น Qubit IQ Assay ไม่เพียงสะดวกในการใช้งานและใช้เวลาน้อยลง (ภายในห้านาที) แต่ยังมีความสัมพันธ์ที่ดีระหว่างค่า IQ ของพารามิเตอร์ที่วัดได้และคุณภาพข้อมูลของการทดลองขั้นปลาย
รูปที่ 5 มีความสัมพันธ์ที่ดีระหว่างค่า Qubit RNA IQ และค่าที่อ่านได้ของ RNA-Seq【5】
จากการแนะนำข้างต้น ผมเชื่อว่าทุกคนมีความเข้าใจเพียงพอเกี่ยวกับวิธีการควบคุมคุณภาพ RNA ที่แตกต่างกันในทางปฏิบัติคุณสามารถเลือกได้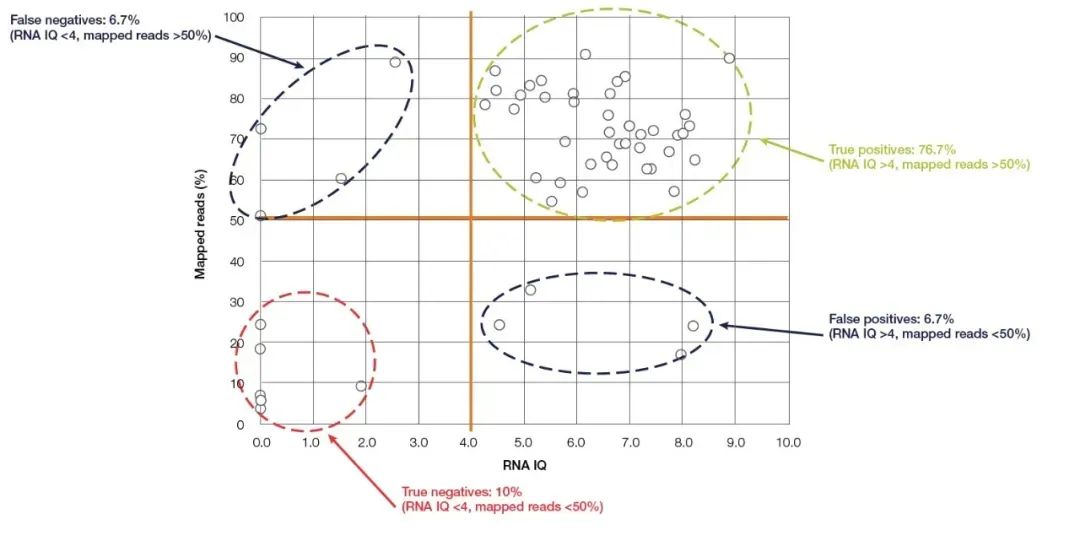 วิธีการที่สอดคล้องกับประเภทตัวอย่างและเครื่องมือที่มีอยู่การควบคุมคุณภาพของ RNA ให้ดีเท่านั้นที่จะทำให้เราหลีกเลี่ยงความล้มเหลวของการทดลองที่ตามมาซึ่งเกิดจากตัวอย่างที่มีคุณภาพต่ำ ซึ่งช่วยประหยัดเวลาอันมีค่า พลังงาน และค่าใช้จ่าย
วิธีการที่สอดคล้องกับประเภทตัวอย่างและเครื่องมือที่มีอยู่การควบคุมคุณภาพของ RNA ให้ดีเท่านั้นที่จะทำให้เราหลีกเลี่ยงความล้มเหลวของการทดลองที่ตามมาซึ่งเกิดจากตัวอย่างที่มีคุณภาพต่ำ ซึ่งช่วยประหยัดเวลาอันมีค่า พลังงาน และค่าใช้จ่าย
สินค้าอ้างอิง:
อ้างอิง
【1】Schroeder, A., Mueller, O., Stocker, S. และคณะRIN: หมายเลขความสมบูรณ์ของ RNA สำหรับการกำหนดค่าความสมบูรณ์ให้กับการวัด RNABMC ชีวโมเลกุล 7, 3 (2549)https:// doi .org/10.1186/1471-21 99-7-3
【2】คู่มือผู้ใช้โปรแกรมสร้างภูมิคุ้มกันของมนุษย์เมื่อมาถึง (Pub. No. MAN0017438 Rev. C.0)
【3】Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, เมตริกคุณภาพที่ได้รับการปรับปรุงสำหรับการประเมิน RNA ที่ได้มาจากตัวอย่างเนื้อเยื่อฝังพาราฟินคงที่ฟอร์มาลิน, วิทยาศาสตร์พิษวิทยา, เล่มที่ 170, ฉบับที่ 2, สิงหาคม 2019, หน้า 357–373,https://doi.org/10.1093/toxsci/
เวลาโพสต์: Jun-12-2023