ความจำเพาะของการตรวจจับ
ในกรณีส่วนใหญ่ จุดประสงค์ของการออกแบบไพรเมอร์คือเพื่อเพิ่มลักษณะเฉพาะของ PCRสิ่งนี้ถูกกำหนดโดยอิทธิพลที่คาดเดาได้มากหรือน้อยของตัวแปรหลายตัวตัวแปรสำคัญอย่างหนึ่งคือลำดับที่ 3′end ของไพรเมอร์
ที่สำคัญ การทดสอบ PCR ที่ออกแบบมาสำหรับความเฉพาะเจาะจงมีแนวโน้มที่จะรักษาประสิทธิภาพสูงในช่วงไดนามิกที่กว้าง เนื่องจากการทดสอบไม่ได้ผลิตผลิตภัณฑ์ขยายสัญญาณที่ไม่เฉพาะเจาะจง ดังนั้นจึงแข่งขันกับ PCR reagents หรือยับยั้งปฏิกิริยาการขยายสัญญาณหลัก
แน่นอน ในบางกรณี ความเฉพาะเจาะจงไม่ใช่สิ่งที่สำคัญที่สุด ตัวอย่างเช่น เมื่อเป้าหมายคือการหาปริมาณของเชื้อโรคที่เกี่ยวข้องกันอย่างใกล้ชิดแต่ต่างกัน จำเป็นต้องมีการออกแบบพิเศษ การเพิ่มประสิทธิภาพ และมาตรฐานการตรวจสอบ
เส้นโค้งการหลอมละลายเป็นวิธีการมาตรฐานสำหรับการประเมินความจำเพาะของแอมพลิคอน อย่างน้อยก็ในแง่ของการขยายเป้าหมายเดียวหรือไม่อย่างไรก็ตาม ต้องเน้นย้ำว่ากราฟการหลอมละลายอาจทำให้เข้าใจผิดได้ เพราะตัวอย่างเช่น อาจได้รับผลกระทบจากผลรวมของไพรเมอร์ที่ไม่เหมาะสมและความเข้มข้นของแม่แบบที่ต่ำ
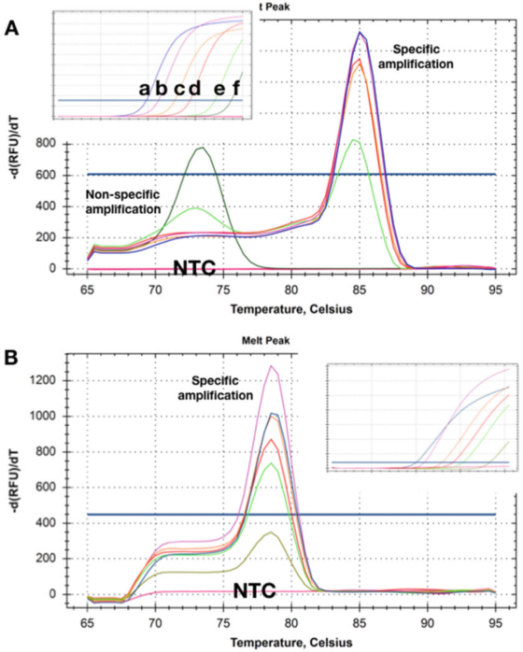
P5 |เส้นโค้งการหลอมเหลวแสดงการเปลี่ยนแปลงของ Tm ที่ได้จากการตรวจจับสองครั้งของ DNA เป้าหมายสองตัวในปริมาณที่แตกต่างกัน
A. ที่ความเข้มข้นสูงกว่า (โฆษณา)) จะไม่มีไพรเมอร์ไดเมอร์ที่ชัดเจนหลังจากการตรวจวัด qPCR เสร็จสิ้นเมื่อความเข้มข้นของเทมเพลตลดลงเหลือ 50 สำเนา (e) ผลิตภัณฑ์ที่ไม่เฉพาะเจาะจงจะเริ่มปรากฏขึ้นและกลายเป็นผลิตภัณฑ์เดียวที่ความเข้มข้นต่ำสุด (f)
B. การทดสอบบันทึกค่า Tms เท่ากันที่ความเข้มข้นเป้าหมายทั้งหมด และไม่มีไพรเมอร์ไดเมอร์ที่ชัดเจนแม้ที่ความเข้มข้นต่ำสุด (5 ชุด)เมื่อใช้วิธีการตรวจจับทั้งสองนี้ จะไม่พบผลิตภัณฑ์ขยายเสียงใน NTC
P5 แสดงเส้นโค้งการละลายที่ได้จากตัวอย่างซึ่งมีแม่แบบอยู่ที่ความเข้มข้นต่างกันP 5a แสดงให้เห็นว่าที่ความเข้มข้นต่ำสุดสองค่า Tms ของผลิตภัณฑ์แอมพลิฟายเออร์ที่ไม่เฉพาะเจาะจงที่ผลิตได้นั้นต่ำกว่าแอมพลิฟายเออร์เฉพาะ
เห็นได้ชัดว่าวิธีการตรวจจับนี้ไม่สามารถใช้เพื่อตรวจจับเป้าหมายที่มีความเข้มข้นต่ำได้อย่างน่าเชื่อถือ
ที่น่าสนใจคือ กทช เช่น ตัวอย่างที่ไม่มี DNA เลย ไม่ได้บันทึกผลิตภัณฑ์การขยาย (ไม่เฉพาะเจาะจง) ซึ่งบ่งชี้ว่า DNA ของจีโนมพื้นหลังสามารถมีส่วนร่วมในการขยาย/พอลิเมอไรเซชันที่ไม่เฉพาะเจาะจงได้
บางครั้งไพรเมอร์พื้นหลังและการขยายที่ไม่เฉพาะเจาะจงไม่สามารถแก้ไขได้ แต่บ่อยครั้งก็เป็นไปได้ที่จะออกแบบวิธีการตรวจจับที่ไม่มีการขยายที่ไม่เฉพาะเจาะจงในความเข้มข้นของแม่แบบและ NTC (P 5b)
ที่นี่ แม้แต่การบันทึกการขยายความเข้มข้นของเป้าหมายด้วย Cq ที่ 35 ก็จะสร้างเส้นโค้งการละลายที่เฉพาะเจาะจงในทำนองเดียวกัน กทช. ไม่พบสัญญาณของการขยายสัญญาณที่ไม่เฉพาะเจาะจงบางครั้ง พฤติกรรมการตรวจจับอาจขึ้นอยู่กับเหล้าแม่ และตรวจพบเฉพาะการขยายที่ไม่เฉพาะเจาะจงในองค์ประกอบบัฟเฟอร์บางอย่าง ซึ่งอาจเกี่ยวข้องกับความเข้มข้นของ Mg2+ ที่แตกต่างกัน
ความเสถียรของการตรวจจับ
การปรับให้เหมาะสมของ Ta เป็นขั้นตอนที่มีประโยชน์ในการตรวจสอบเชิงประจักษ์และกระบวนการปรับให้เหมาะสมของการตรวจจับ qPCRให้ตัวบ่งชี้โดยตรงของความทนทานของสีรองพื้นที่ตั้งไว้โดยการแสดงอุณหภูมิ (หรือช่วงอุณหภูมิ) ที่สร้าง Cq ต่ำสุดโดยไม่ต้องขยาย NTC
ความแตกต่างของความไวสองถึงสี่เท่าอาจไม่สำคัญสำหรับผู้ที่มีการแสดงออกของ mRNA สูง แต่สำหรับการตรวจวินิจฉัย อาจหมายถึงความแตกต่างระหว่างผลบวกและผลลบที่ผิดพลาด
คุณสมบัติ Ta ของไพรเมอร์ qPCR อาจแตกต่างกันอย่างมากการทดสอบบางอย่างไม่มีประสิทธิภาพมากนัก และหากไม่ได้ดำเนินการภายใต้ค่า Ta ที่เหมาะสมที่สุดของไพรเมอร์ การทดสอบเหล่านั้นจะพังทลายลงอย่างรวดเร็ว
นี่เป็นสิ่งสำคัญเนื่องจากการตรวจจับประเภทนี้มักเป็นปัญหาในโลกแห่งความเป็นจริง และความบริสุทธิ์ของตัวอย่าง ความเข้มข้นของ DNA หรือการมีอยู่ของ DNA อื่นๆ อาจไม่เหมาะสม
นอกจากนี้ หมายเลขสำเนาเป้าหมายอาจแตกต่างกันไปในช่วงกว้าง และน้ำยา เครื่องใช้พลาสติก หรือเครื่องมืออาจแตกต่างจากที่ใช้เมื่อตั้งค่าการทดสอบ
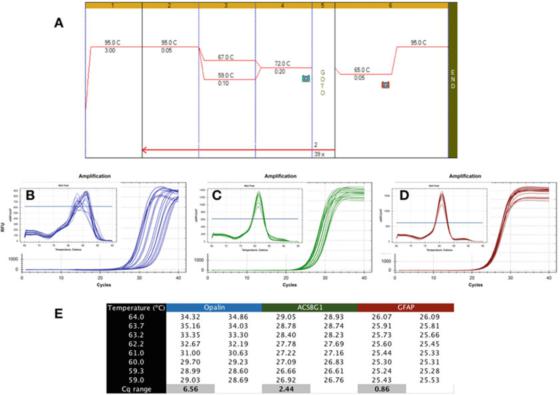
P6|การไล่ระดับอุณหภูมิแสดงถึงความทนทานที่แตกต่างกันของการตรวจจับ PCR
A. ใช้ Sensifast SYBR mastermix ของ Bioline (หมายเลขแค็ตตาล็อก BIO-98050) เพื่อทำ PCR บน cDNA ที่เตรียมจาก RNA ของสมองมนุษย์
B. ใช้เครื่องมือ CFX qPCR ของ Bio-Rad เพื่อบันทึกแผนผังการขยายและเส้นโค้งการละลายของ apalene (NM_033207, F: GCCATGGAGGAAAAGTGACAGACC, R: CTCATGTGTGGGTGTGATCTCCTAGG)
C. กราฟการขยายและกราฟการหลอมละลายของ ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTTGTCTTGACTCAG)
D. กราฟการขยายและเส้นโค้งการละลายของ GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCCGCTGG, R: CGAACCTCCTCCTCCGTGGATCTCTC)
E. ค่า Cq ที่บันทึกที่อุณหภูมิการหลอมต่างกัน แสดงค่าความแตกต่างของค่า Cq ที่บันทึกภายใต้การไล่ระดับอุณหภูมิ 7 องศาเซลเซียส
P 6 แสดงผลโดยทั่วไปของการทดสอบที่ไม่พึงประสงค์ โดยทำ qPCR โดยใช้การไล่ระดับสี Tas ระหว่าง 59C และ 67C (P 6a) โดยใช้ไพรเมอร์สำหรับยีนเฉพาะสมองของมนุษย์สามตัว
เห็นได้จากกราฟแอมพลิฟายเออร์ว่าไพรเมอร์ Opalin นั้นห่างไกลจากอุดมคติ เนื่องจากช่วง Ta ที่เหมาะสมที่สุดของพวกมันนั้นแคบมาก (รูปที่ 6b) นั่นคือ Cqs กระจายตัวอย่างกว้างขวาง ส่งผลให้ Cqs ถูกเปรียบเทียบอย่างมีนัยสำคัญกับ Cqs ต่ำที่เหมาะสมที่สุด
วิธีการตรวจจับนี้ไม่เสถียรและอาจนำไปสู่การขยายที่ต่ำกว่ามาตรฐานดังนั้นควรออกแบบไพรเมอร์คู่นี้ใหม่นอกจากนี้ การวิเคราะห์เส้นโค้งการหลอมเหลว (สิ่งที่ใส่เข้าไป) แสดงให้เห็นว่าความจำเพาะของวิธีการตรวจจับนี้อาจเป็นปัญหาได้เช่นกัน เนื่องจากเส้นโค้งการหลอมละลายของ Ta แต่ละชนิดนั้นแตกต่างกัน
วิธีการตรวจจับ ACSBG1 ที่แสดงใน P 6c นั้นแข็งแกร่งกว่าวิธีการตรวจจับ Opalin ด้านบน แต่ก็ยังห่างไกลจากอุดมคติ และมีแนวโน้มว่าจะสามารถปรับปรุงได้
อย่างไรก็ตาม เราเน้นย้ำว่าไม่มีความเชื่อมโยงที่จำเป็นระหว่างความทนทานและความจำเพาะ เนื่องจากเส้นโค้งการละลายที่ผลิตโดยวิธีการตรวจจับนี้แสดงค่าสูงสุดเดียวกันในทุก Tas (สิ่งที่ใส่เข้าไป)
ในทางกลับกัน การทดสอบความทนทานมีความทนทานมากกว่ามาก โดยสร้าง Cqs ที่คล้ายกันใน Tas ที่หลากหลาย เช่นเดียวกับในการทดสอบ GFAP ที่แสดงใน P 6d
ความแตกต่างของ Cqs ที่ได้รับในช่วง 8 องศาเซลเซียสเดียวกันนั้นน้อยกว่า 1 และกราฟการละลาย (ภาพประกอบ) ยืนยันลักษณะการตรวจจับในช่วงอุณหภูมินี้เป็นที่น่าสังเกตว่า Tas ที่คำนวณได้และช่วง Ta จริงอาจแตกต่างกันมาก
มีแนวทางมากมายที่ออกแบบมาเพื่อช่วยให้นักวิจัยออกแบบไพรเมอร์ที่มีประสิทธิภาพ ซึ่งส่วนใหญ่เป็นไปตามกฎที่มีมายาวนานมักจะแนะนำให้รวม G หรือ C ที่ปลาย 3' และฐาน G หรือ C สองฐาน (แคลมป์ GC) แต่ไม่เกินสองฐานจาก 5 ฐานสุดท้าย
ในทางปฏิบัติ กฎเหล่านี้สามารถเป็นแนวทางให้นักวิจัยได้ แต่ไม่จำเป็นต้องถูกต้องในทุกสถานการณ์
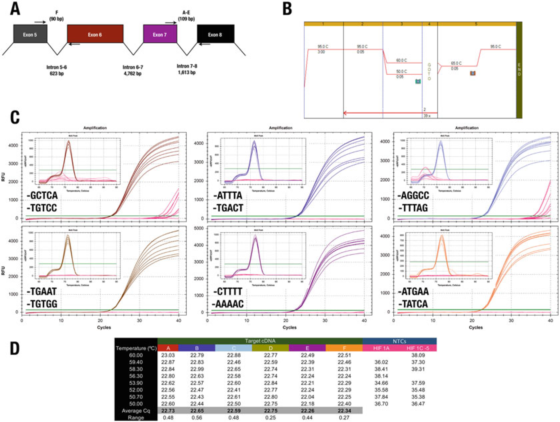
P7 |3′end ของไพรเมอร์มีผลเพียงเล็กน้อยต่อความจำเพาะหรือประสิทธิภาพ
A. ตำแหน่งของไพรเมอร์สำหรับยีน HIF-1α (NM_181054.2) ของมนุษย์
B. ใช้ Agilent Brilliant III SYBR Green Mother Liquor (Cat. No. 600882) เพื่อขยายรายการทดสอบหกรายการ
C. กราฟการขยายและกราฟการหลอมละลายที่บันทึกโดยเครื่องมือ CFX qPCR ของ Bio-Rad และไพรเมอร์ 3′endNTC แสดงเป็นสีแดง
D. บันทึก Cqs ของแต่ละรายการทดสอบ
ตัวอย่างเช่น ผลลัพธ์ใน P 7 ขัดแย้งกับกฎ 3′endการออกแบบทั้งหมดให้ผลลัพธ์ที่เหมือนกัน โดยพื้นฐานแล้วมีเพียงการผสมไพรเมอร์สองชุดเท่านั้นที่นำไปสู่การขยายที่ไม่เฉพาะเจาะจงใน NTC
อย่างไรก็ตาม เราไม่สามารถสนับสนุนเอฟเฟกต์ของคลิป GC ได้ เนื่องจากในกรณีนี้ การใช้ A หรือ T สูงสุด 30 ฐานไม่ได้ลดความจำเพาะ
ทดสอบ C โดยที่ไพรเมอร์ F ลงท้ายด้วย GGCC บันทึก Cqs ใน NTC ซึ่งบ่งชี้ว่าอาจต้องการหลีกเลี่ยงลำดับเหล่านี้ที่ปลาย 30เราเน้นย้ำว่าวิธีเดียวที่จะกำหนดลำดับ 3′end ที่ดีที่สุดของคู่ไพรเมอร์คือการประเมินตัวเลือกไพรเมอร์บางตัวด้วยการทดลอง
ประสิทธิภาพการขยาย
สิ่งสำคัญ แม้ว่าการตรวจจับ PCR แบบไม่เฉพาะเจาะจงจะไม่สามารถเจาะจงได้ แต่ประสิทธิภาพการขยายสัญญาณสามารถปรับและขยายสูงสุดได้หลายวิธีโดยการเปลี่ยนเอนไซม์ เหล้าแม่ สารเติมแต่ง และสภาวะการวนรอบ
ในการประเมินประสิทธิภาพของการตรวจจับ PCR วิธีที่ดีที่สุดคือใช้การเจือจางแบบอนุกรม 10 หรือ 5 เท่าของกรดนิวคลีอิกเป้าหมาย ซึ่งก็คือ "วิธีเส้นโค้งมาตรฐาน"
ถ้าใช้ PCR amplicons หรือ DNA เป้าหมายสังเคราะห์เพื่อสร้างเส้นโค้งมาตรฐาน ควรผสมการเจือจางแบบอนุกรมของเป้าหมายเหล่านี้กับ DNA พื้นหลังในปริมาณคงที่ (เช่น DNA จีโนม)
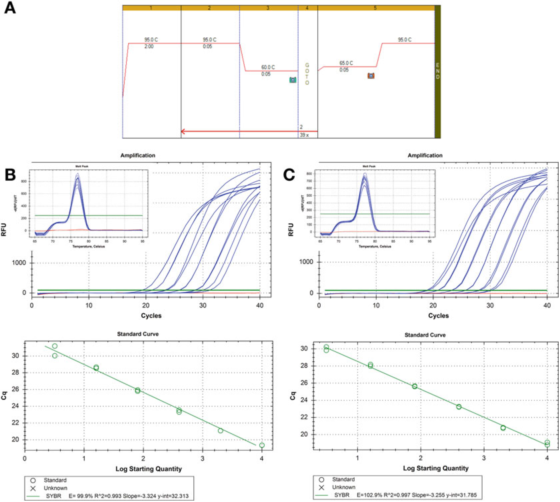
P8 |เส้นโค้งการเจือจางเพื่อประเมินประสิทธิภาพของ PCR
A. ใช้ไพรเมอร์สำหรับ HIF-1: F: AAGAACTTTTAGGCCGCTCA และ R: TGTCCTTGTGGTGACTTGTCC และ Agilent's Brilliant III SYBR Green mastermix (หมายเลขแคตตาล็อก 600882) สำหรับ PCR และสภาวะเส้นโค้งการหลอมเหลว
B. 100 ng RNA ถูกคัดลอกแบบย้อนกลับ เจือจาง 2 ครั้ง และตัวอย่าง cDNA ที่เจือจางแบบอนุกรมถูกเจือจาง 5 ครั้งเป็น 1 ng DNA ของจีโนมมนุษย์เส้นโค้งการหลอมละลายแสดงอยู่ในสิ่งที่ใส่เข้าไป
C. ปฏิกิริยา RT การเจือจาง และการเจือจางแบบอนุกรมถูกทำซ้ำสำหรับตัวอย่าง cDNA ที่สอง และผลลัพธ์ก็คล้ายกัน
P 8 แสดงเส้นโค้งมาตรฐานสองเส้น โดยใช้วิธีการตรวจจับเดียวกันในตัวอย่าง cDNA สองตัวอย่างที่ต่างกัน ผลลัพธ์คือประสิทธิภาพเท่ากันประมาณ 100% และค่า R2 ก็ใกล้เคียงกัน นั่นคือระดับความพอดีระหว่างข้อมูลการทดลองกับเส้นถดถอยหรือระดับของความเป็นเส้นตรงของข้อมูล
เส้นโค้งมาตรฐานทั้งสองนั้นเปรียบเทียบกันได้ แต่ไม่เหมือนกันทุกประการหากมีวัตถุประสงค์เพื่อวัดปริมาณเป้าหมายอย่างถูกต้อง จะต้องสังเกตว่าไม่สามารถให้การคำนวณหมายเลขสำเนาโดยไม่อธิบายความไม่แน่นอนได้
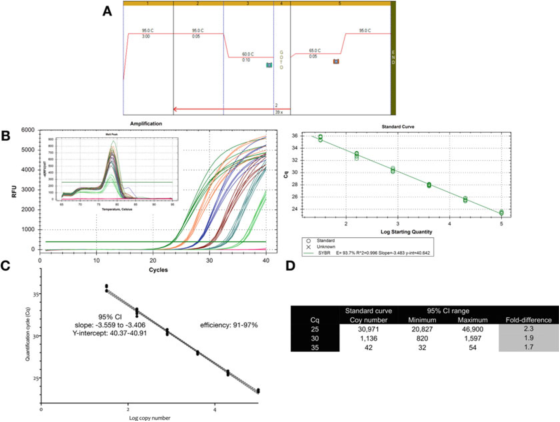
P9 |ความไม่แน่นอนของการวัดที่เกี่ยวข้องกับการหาปริมาณโดยใช้เส้นโค้งมาตรฐาน.
A. ใช้ไพรเมอร์สำหรับ GAPDH (NM_002046) เพื่อดำเนินการ PCR และสภาวะเส้นโค้งการหลอมเหลวF: ACAGTTGCCATGTAGACC และ R: TAACTGGTTGAGCACAGG และ Sensifast SYBR mastermix ของ Bioline (หมายเลขแค็ตตาล็อก BIO-98050)
B. แผนภูมิการขยาย เส้นโค้งการหลอมเหลว และเส้นโค้งมาตรฐานที่บันทึกด้วยเครื่องมือ CFX qPCR ของ Bio-Rad
ค. กราฟเส้นโค้งมาตรฐานและช่วงความเชื่อมั่น 95% (CI).
D. จำนวนสำเนาและช่วงความเชื่อมั่น 95% ของค่า Cq สามค่าที่ได้มาจากเส้นโค้งการเจือจาง
P 9 แสดงให้เห็นว่าสำหรับการทดสอบที่ปรับให้เหมาะสม ความแปรปรวนโดยธรรมชาติของเส้นโค้งมาตรฐานเดียวคือประมาณ 2 เท่า (ช่วงความเชื่อมั่น 95% ต่ำสุดถึงสูงสุด) ซึ่งอาจเป็นความแปรปรวนที่น้อยที่สุดที่สามารถคาดหวังได้
สินค้าที่เกี่ยวข้อง:
เวลาโพสต์: กันยายน 30-2021