1. ความรู้พื้นฐาน (หากต้องการดูส่วนทดลอง โปรดโอนโดยตรงไปยังส่วนที่สอง)
ในฐานะที่เป็นปฏิกิริยาอนุพันธ์ของ PCR แบบเดิม PCR แบบเรียลไทม์จะตรวจสอบการเปลี่ยนแปลงของปริมาณของผลิตภัณฑ์การขยายในแต่ละรอบของปฏิกิริยาการขยาย PCR แบบเรียลไทม์ผ่านการเปลี่ยนแปลงของสัญญาณเรืองแสงเป็นหลัก และวิเคราะห์แม่แบบเริ่มต้นเชิงปริมาณผ่านความสัมพันธ์ระหว่างค่า ct และเส้นโค้งมาตรฐาน
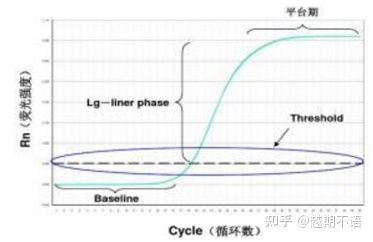
ข้อมูลเฉพาะของ RT-PCR ได้แก่พื้นฐาน, เกณฑ์การเรืองแสงและค่ากะรัต
| พื้นฐาน: | ค่าฟลูออเรสเซนต์ของรอบที่ 3-15 คือค่าพื้นฐาน (baseline) ซึ่งเกิดจากความผิดพลาดของการวัดเป็นครั้งคราว |
| เกณฑ์ (เกณฑ์): | หมายถึงขีดจำกัดการตรวจจับการเรืองแสงที่ตำแหน่งที่เหมาะสมในบริเวณการเติบโตแบบทวีคูณของเส้นโค้งการขยาย โดยทั่วไปคือ 10 เท่าของค่าเบี่ยงเบนมาตรฐานของเส้นฐาน |
| ค่า CT: | เป็นจำนวนรอบ PCR เมื่อค่าการเรืองแสงในแต่ละหลอดปฏิกิริยาถึงเกณฑ์ ค่า Ct จะแปรผกผันกับจำนวนเทมเพลตเริ่มต้น |
วิธีการติดฉลากทั่วไปสำหรับ RT-PCR:
| วิธี | ข้อได้เปรียบ | ข้อบกพร่อง | ขอบเขตการใช้งาน |
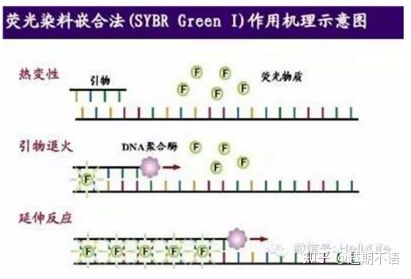
| SYBR สีเขียวⅠ | การบังคับใช้กว้าง ละเอียดอ่อน ราคาถูกและสะดวก | ความต้องการสีรองพื้นสูง มีแนวโน้มที่จะเกิดแถบสีที่ไม่เฉพาะเจาะจง | เหมาะสำหรับการวิเคราะห์เชิงปริมาณของยีนเป้าหมายต่างๆ การวิจัยเกี่ยวกับการแสดงออกของยีน และการวิจัยเกี่ยวกับสัตว์และพืชดัดแปลงพันธุกรรม |
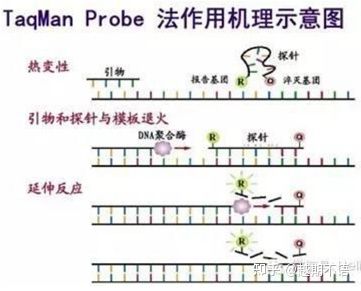
| แท็กแมน | ความจำเพาะที่ดีและความสามารถในการทำซ้ำสูง | ราคาสูงและเหมาะสำหรับเป้าหมายเฉพาะเท่านั้น | การตรวจหาเชื้อก่อโรค การวิจัยยีนดื้อยา การประเมินประสิทธิภาพของยา การวินิจฉัยโรคทางพันธุกรรม |
| สัญญาณโมเลกุล | ความจำเพาะสูง การเรืองแสง พื้นหลังต่ำ | ราคาสูง เหมาะสำหรับวัตถุประสงค์เฉพาะ การออกแบบยาก และราคาสูง | การวิเคราะห์ยีนเฉพาะ การวิเคราะห์ SNP |
2. ขั้นตอนการทดลอง
2.1 เกี่ยวกับการจัดกลุ่มทดลอง- ต้องมีหลายหลุมในกลุ่มและต้องมีการทำซ้ำทางชีวภาพ
| ① | การควบคุมที่ว่างเปล่า | ใช้ในการตรวจสอบสถานะการเติบโตของเซลล์ในการทดลอง |
| ② | siRNA ควบคุมเชิงลบ (ลำดับ siRNA ที่ไม่เฉพาะเจาะจง) | แสดงให้เห็นถึงความเฉพาะเจาะจงของการกระทำของ RNAisiRNA อาจกระตุ้นการตอบสนองต่อความเครียดที่ไม่เฉพาะเจาะจงที่ความเข้มข้น 200nM |
| ③ | การควบคุมรีเอเจนต์ทรานส์เฟกชัน | ไม่รวมความเป็นพิษของสารทำปฏิกิริยาการเปลี่ยนถ่ายต่อเซลล์หรือผลกระทบต่อการแสดงออกของยีนเป้าหมาย |
| ④ | siRNA กับยีนเป้าหมาย | ทำให้การแสดงออกของยีนเป้าหมายลดลง |
| ⑤ (ไม่บังคับ) | siRNA เชิงบวก | ใช้เพื่อแก้ไขปัญหาระบบการทดลองและปัญหาการปฏิบัติงาน |
| ⑥ (ไม่บังคับ) | siRNA ควบคุมฟลูออเรสเซนต์ | ประสิทธิภาพของการถ่ายเซลล์สามารถสังเกตได้ด้วยกล้องจุลทรรศน์ |
2.2 หลักการออกแบบสีรองพื้น
| ขยายขนาดชิ้นส่วน | โดยเฉพาะอย่างยิ่งที่ 100-150bp |
| ความยาวของไพรเมอร์ | 18-25bp |
| เนื้อหา GC | 30%-70% โดยเฉพาะอย่างยิ่ง 45%-55% |
| ค่าทีเอ็ม | 58-60 ℃ |
| ลำดับ | หลีกเลี่ยง T/C อย่างต่อเนื่องA/G ต่อเนื่อง |
| 3 ลำดับท้าย | หลีกเลี่ยง GC รวยหรือ AT รวยฐานขั้วต่ออย่างพึงประสงค์คือ G หรือ C;ทางที่ดีควรหลีกเลี่ยง T |
| เสริม | หลีกเลี่ยงลำดับเบสเสริมมากกว่า 3 เบสภายในไพรเมอร์หรือระหว่างไพรเมอร์สองตัว |
| ความเฉพาะเจาะจง | ใช้การค้นหาด้วยระเบิดเพื่อยืนยันความจำเพาะของไพรเมอร์ |
①SiRNA มีลักษณะเฉพาะของสปีชีส์ และลำดับของสปีชีส์ต่างๆ จะแตกต่างกัน
②SiRNA บรรจุในผงแห้งเยือกแข็ง ซึ่งสามารถเก็บไว้ได้อย่างเสถียรเป็นเวลา 2-4 สัปดาห์ที่อุณหภูมิห้อง
2.3 เครื่องมือหรือน้ำยาที่ต้องเตรียมล่วงหน้า
| สีรองพื้น (อ้างอิงภายใน) | รวมถึงไปข้างหน้าและย้อนกลับสอง |
| ไพรเมอร์ (ยีนเป้าหมาย) | รวมถึงไปข้างหน้าและย้อนกลับสอง |
| เป้าหมาย Si RNA (3 แถบ) | โดยทั่วไป บริษัทจะสังเคราะห์ 3 แถบ แล้วเลือกหนึ่งในสามโดย RT-PCR |
| ชุดเปลี่ยนถ่าย | ไลโป2000เป็นต้น |
| ชุดสกัดอย่างรวดเร็วของ RNA | สำหรับการสกัด RNA หลังจากการถ่ายเซลล์ |
| ชุดถอดความแบบย้อนกลับอย่างรวดเร็ว | สำหรับการสังเคราะห์ cDNA |
| ชุดขยายสัญญาณ PCR | 2×Super SYBR สีเขียว qPCR มาสเตอร์มิกซ์ |
2.4 เกี่ยวกับประเด็นที่ต้องให้ความสนใจในขั้นตอนการทดลองเฉพาะ:
①กระบวนการเปลี่ยนถ่าย SiRNA
1. สำหรับการชุบ คุณสามารถเลือกเพลต 24 หลุม, เพลต 12 หลุม หรือเพลต 6 หลุม (ความเข้มข้นของ RNA เฉลี่ยที่เสนอในแต่ละหลุมของเพลต 24 หลุมคือประมาณ 100-300 ng/uL) และความหนาแน่นของการทรานส์เฟกชันที่เหมาะสมของเซลล์สูงถึง 60 %-80% หรือมากกว่านั้น
2. ขั้นตอนการถ่ายและข้อกำหนดเฉพาะเป็นไปตามคำแนะนำอย่างเคร่งครัด
3. หลังจากการถ่ายเซลล์ สามารถเก็บตัวอย่างภายใน 24-72 ชั่วโมงเพื่อตรวจหา mRNA (RT-PCR) หรือตรวจหาโปรตีนภายใน 48-96 ชั่วโมง (WB)
② กระบวนการสกัด RNA
1. ป้องกันการปนเปื้อนจากเอนไซม์ภายนอกรวมถึงการสวมหน้ากากอนามัยและถุงมืออย่างเคร่งครัดการใช้ทิปปิเปตที่ผ่านการฆ่าเชื้อและหลอด EPน้ำที่ใช้ในการทดลองต้องปราศจากอาร์เนส
2. แนะนำให้ทำสองครั้งตามคำแนะนำในชุดการสกัดอย่างรวดเร็ว ซึ่งจะช่วยเพิ่มความบริสุทธิ์และผลผลิตได้อย่างแท้จริง
3. ของเหลวของเสียต้องไม่สัมผัสกับคอลัมน์ RNA
③ ปริมาณ RNA
หลังจากสกัด RNA แล้ว สามารถวัดปริมาณได้โดยตรงด้วย Nanodrop และค่าต่ำสุดที่อ่านได้อาจต่ำถึง 10ng/ul
④ย้อนกลับขั้นตอนการถอดความ
1. เนื่องจาก RT-qPCR มีความไวสูง จึงควรสร้างหลุมขนานอย่างน้อย 3 หลุมสำหรับแต่ละตัวอย่าง เพื่อป้องกันไม่ให้ Ct ที่ตามมาแตกต่างกันมากเกินไป หรือ SD มีขนาดใหญ่เกินไปสำหรับการวิเคราะห์ทางสถิติ
2. ห้ามแช่แข็งและละลายมาสเตอร์มิกซ์ซ้ำๆ
3. ต้องเปลี่ยนท่อ/รูแต่ละอันด้วยปลายอันใหม่!อย่าใช้ปิเปตทิปเดิมอย่างต่อเนื่องเพื่อเพิ่มตัวอย่าง!
4. ฟิล์มที่ติดอยู่บนเพลต 96 หลุมหลังจากใส่ตัวอย่างแล้วจะต้องทำให้เรียบด้วยเพลททางที่ดีควรปั่นแยกก่อนวางบนเครื่อง เพื่อให้ของเหลวที่ผนังท่อไหลลงมาและไล่ฟองอากาศออก
⑤การวิเคราะห์เส้นโค้งทั่วไป
| ไม่มีช่วงของการเติบโตของลอการิทึม | อาจมีเทมเพลตที่มีความเข้มข้นสูง |
| ไม่มีค่า CT | ขั้นตอนไม่ถูกต้องในการตรวจจับสัญญาณเรืองแสง การเสื่อมสภาพของไพรเมอร์หรือโพรบ – สามารถตรวจจับความสมบูรณ์ของมันได้ด้วย PAGE อิเล็กโตรโฟรีซิส จำนวนเทมเพลตไม่เพียงพอ การเสื่อมสภาพของแม่แบบ – หลีกเลี่ยงการนำสิ่งเจือปน และการแช่แข็งและการละลายซ้ำๆ ในการเตรียมตัวอย่าง |
| กะรัต>38 | ประสิทธิภาพการขยายต่ำผลิตภัณฑ์ PCR ยาวเกินไปส่วนประกอบของปฏิกิริยาต่างๆ จะลดลง |
| เส้นโค้งการขยายเชิงเส้น | โพรบอาจเสื่อมสภาพบางส่วนจากรอบการละลายน้ำแข็งซ้ำๆ หรือการเปิดรับแสงเป็นเวลานาน |
| ความแตกต่างของรูที่ซ้ำกันนั้นมีมากเป็นพิเศษ | สารละลายปฏิกิริยาไม่ละลายอย่างสมบูรณ์หรือไม่ผสมสารละลายปฏิกิริยาอ่างความร้อนของเครื่องมือ PCR ปนเปื้อนด้วยสารเรืองแสง |
2.5 เกี่ยวกับการวิเคราะห์ข้อมูล
การวิเคราะห์ข้อมูลของ qPCR สามารถแบ่งออกเป็นปริมาณสัมพัทธ์และปริมาณสัมบูรณ์ตัวอย่างเช่น เซลล์ในกลุ่มการรักษาเปรียบเทียบกับเซลล์ในกลุ่มควบคุม
จำนวนครั้งที่ mRNA ของยีน X เปลี่ยนแปลง นี่คือปริมาณสัมพัทธ์ในเซลล์จำนวนหนึ่ง mRNA ของยีน X
มีกี่ชุด นี่คือปริมาณที่แน่นอนโดยปกติแล้วสิ่งที่เราใช้มากที่สุดในห้องปฏิบัติการคือวิธีการเชิงปริมาณสัมพัทธ์โดยปกติ,วิธี 2-ΔΔctถูกใช้มากที่สุดในการทดลอง ดังนั้นเราจะแนะนำรายละเอียดเฉพาะวิธีการนี้ที่นี่
วิธี 2-ΔΔct: ผลลัพธ์ที่ได้คือความแตกต่างของการแสดงออกของยีนเป้าหมายในกลุ่มทดลองที่สัมพันธ์กับยีนเป้าหมายในกลุ่มควบคุมจำเป็นต้องให้ประสิทธิภาพการขยายของทั้งยีนเป้าหมายและยีนอ้างอิงภายในมีค่าใกล้เคียง 100% และค่าเบี่ยงเบนสัมพัทธ์ไม่ควรเกิน 5%
วิธีการคำนวณมีดังนี้:
Δct กลุ่มควบคุม = ค่า ct ของยีนเป้าหมายในกลุ่มควบคุม – ค่า ct ของยีนอ้างอิงภายในในกลุ่มควบคุม
Δct กลุ่มทดลอง = ค่า ct ของยีนเป้าหมายในกลุ่มทดลอง – ค่า ct ของยีนอ้างอิงภายในในกลุ่มทดลอง
ΔΔct=Δct กลุ่มทดลอง-Δct กลุ่มควบคุม
สุดท้าย คำนวณผลคูณของผลต่างในระดับนิพจน์:
เปลี่ยน Fold=2-ΔΔct (ตรงกับฟังก์ชัน excel คือ POWER)
สินค้าที่เกี่ยวข้อง:
เวลาโพสต์: พฤษภาคม 20-2023