หลังจากที่ Eric S. Lander นักวิชาการชาวอเมริกันเสนอ single nucleotide polymorphism (SNP) เป็นเครื่องหมายโมเลกุลรุ่นที่สามอย่างเป็นทางการในปี 1996 SNP ได้ถูกนำไปใช้อย่างแพร่หลายในการวิเคราะห์การเชื่อมโยงลักษณะทางเศรษฐกิจ การสร้างแผนที่ความเชื่อมโยงทางพันธุกรรมทางชีววิทยา และการคัดกรองยีนที่ทำให้เกิดโรคในมนุษย์, การวินิจฉัยและทำนายความเสี่ยงของโรค , การตรวจหายาเฉพาะบุคคล , และสาขาการวิจัยทางชีววิทยาและการแพทย์อื่นๆในด้านการปรับปรุงพันธุ์พืชเศรษฐกิจ การตรวจจับ SNP สามารถรับรู้ถึงการเลือกลักษณะที่ต้องการได้ตั้งแต่เนิ่นๆการเลือกนี้มีลักษณะของความแม่นยำสูงและสามารถหลีกเลี่ยงการรบกวนของลักษณะทางสัณฐานวิทยาและปัจจัยด้านสิ่งแวดล้อมได้อย่างมีประสิทธิภาพ ซึ่งจะทำให้กระบวนการผสมพันธุ์สั้นลงอย่างมากดังนั้น SNP จึงมีบทบาทอย่างมากในด้านการวิจัยขั้นพื้นฐาน
ความแตกต่างของนิวคลีโอไทด์เดี่ยว (Single Nucleotide Polymorphism, SNP) หมายถึง ปรากฏการณ์ที่มีความแตกต่างของนิวคลีโอไทด์เดี่ยวในตำแหน่งเดียวกันในลำดับดีเอ็นเอของบุคคลที่มีสปีชีส์เดียวกันหรือต่างกันการแทรก การลบ การแปลง และการผกผันของฐานเดียวสามารถทำให้เกิดความแตกต่างนี้ได้ในอดีต คำจำกัดความของ SNP แตกต่างจากการกลายพันธุ์ตำแหน่งที่แปรผันต้องการความถี่ของหนึ่งในอัลลีลในประชากรมากกว่า 1% จึงจะกำหนดเป็นโลคัส SNPอย่างไรก็ตาม ด้วยการขยายตัวของทฤษฎีทางชีววิทยาสมัยใหม่และการประยุกต์ใช้เทคโนโลยี ความถี่อัลลีลจึงไม่ใช่เงื่อนไขที่จำเป็นในการจำกัดคำจำกัดความของ SNP อีกต่อไปตามข้อมูลการแปรผันของนิวคลีโอไทด์เดี่ยวที่รวมอยู่ในฐานข้อมูล Single Nucleotide Polymorphisms (dbSNP) ภายใต้ศูนย์ข้อมูลเทคโนโลยีชีวภาพแห่งชาติ (NCBI) การแทรก/การลบความถี่ต่ำ การแปรผันของไมโครแซทเทลไลท์ ฯลฯ รวมอยู่ด้วย
ในร่างกายมนุษย์ ความถี่ของ SNP คือ 0.1%กล่าวอีกนัยหนึ่ง มีไซต์ SNP เฉลี่ยหนึ่งไซต์ต่อ 1,000 คู่เบสแม้ว่าความถี่ของเหตุการณ์จะค่อนข้างสูง แต่ไซต์ SNP บางแห่งเท่านั้นที่สามารถเป็นเครื่องหมายตัวเลือกที่เกี่ยวข้องกับคุณลักษณะได้สิ่งนี้เกี่ยวข้องกับตำแหน่งที่เกิด SNP เป็นหลัก
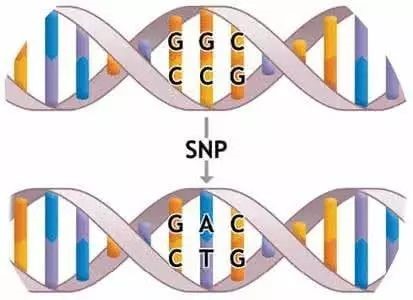
ตามทฤษฎีแล้ว SNP สามารถเกิดขึ้นได้ทุกที่ในลำดับจีโนมSNP ที่เกิดขึ้นในบริเวณการเข้ารหัสสามารถก่อให้เกิดการกลายพันธุ์ที่มีความหมายเหมือนกันและการกลายพันธุ์ที่ไม่มีความหมายเหมือนกัน กล่าวคือ กรดอะมิโนเปลี่ยนแปลงหรือไม่เปลี่ยนแปลงก่อนและหลังการกลายพันธุ์กรดอะมิโนที่เปลี่ยนแปลงมักจะทำให้สายเปปไทด์สูญเสียหน้าที่เดิม (การกลายพันธุ์แบบผิดๆ) และอาจทำให้การแปลผลหยุดชะงัก (การกลายพันธุ์แบบไร้สาระ)SNPs ที่เกิดขึ้นในบริเวณที่ไม่ได้เข้ารหัสและบริเวณระหว่างพันธุกรรมอาจส่งผลต่อการประกบ mRNA, องค์ประกอบลำดับ RNA ที่ไม่ได้เข้ารหัส และประสิทธิภาพการจับของปัจจัยการถอดความและ DNAความสัมพันธ์เฉพาะแสดงในรูป:
ประเภท SNP:
วิธีการพิมพ์ SNP ทั่วไปหลายวิธีและการเปรียบเทียบ
ตามหลักการที่แตกต่างกัน วิธีการตรวจหา SNP ทั่วไปจะแบ่งออกเป็นประเภทต่อไปนี้:
การเปรียบเทียบการจำแนกประเภทของวิธีการตรวจจับ
หมายเหตุ: ปัจจุบันมีการใช้วิธีการตรวจจับ SNP ทั่วไปมากขึ้นตามรายการในตาราง วิธีการตรวจจับอื่นๆ เช่น การไฮบริดไซต์เฉพาะ (ASH) ส่วนขยายไพรเมอร์ไซต์เฉพาะ (ASPE) ส่วนขยายฐานเดี่ยว (SBCE) การตัดไซต์เฉพาะ (ASC) เทคโนโลยีชิปยีน เทคโนโลยีแมสสเปกโตรเมตรี ฯลฯ ยังไม่ได้รับการจัดประเภทและเปรียบเทียบ
ต้นทุนและเวลาในการทำให้กรดนิวคลีอิกบริสุทธิ์ในวิธีการตรวจหา SNP ทั่วไปหลายวิธีข้างต้นเป็นสิ่งที่หลีกเลี่ยงไม่ได้อย่างไรก็ตาม ชุดอุปกรณ์ที่เกี่ยวข้องซึ่งใช้เทคโนโลยี Direct PCR ของ Foregene สามารถดำเนินการขยาย PCR หรือ qPCR ได้โดยตรงกับตัวอย่างที่ไม่บริสุทธิ์ ซึ่งนำความสะดวกสบายอย่างไม่เคยมีมาก่อนมาสู่การตรวจจับ SNP
ผลิตภัณฑ์ซีรีส์ PCR โดยตรงของ Foregene แทบไม่ต้องผ่านขั้นตอนการทำให้บริสุทธิ์ตัวอย่าง ซึ่งช่วยลดเวลาและค่าใช้จ่ายในการเตรียมแม่แบบได้อย่างมากTaq polymerase ที่เป็นเอกลักษณ์มีความสามารถในการขยายสัญญาณที่ยอดเยี่ยมและสามารถทนต่อสารยับยั้งที่หลากหลายจากสภาพแวดล้อมการขยายที่ซับซ้อนคุณลักษณะเหล่านี้รับประกันทางเทคนิคสำหรับการได้รับผลิตภัณฑ์เฉพาะที่ให้ผลผลิตสูง ชุด Foregene Direct PCR/qPCR สำหรับตัวอย่างประเภทต่างๆ เช่น เนื้อเยื่อสัตว์ (หางหนู ปลาม้าลาย ฯลฯ) ใบพืช เมล็ดพืช (รวมถึงตัวอย่างโพลีแซคคาไรด์และโพลีฟีนอล) เป็นต้น
เวลาโพสต์: กรกฎาคม-23-2021