Ⅰ. เพิ่มความไวของระบบปฏิกิริยา:
1. แยก RNA คุณภาพสูง:
การสังเคราะห์ cDNA ที่ประสบความสำเร็จมาจาก RNA คุณภาพสูงRNA คุณภาพสูงควรตรวจสอบให้แน่ใจว่ามีความยาวรวมอย่างน้อยและไม่มีสารยับยั้งที่ไม่มีเอ็นไซม์บันทึก เช่น EDTA หรือ SDSคุณภาพของ RNA จะกำหนดค่าสูงสุดของข้อมูลลำดับที่คุณสามารถถ่ายทอดไปยัง cDNA ได้วิธีการทำให้บริสุทธิ์ RNA ทั่วไปเป็นวิธีขั้นตอนของการใช้ไอโซไซยาเนต/แอซิโดฟีนอลเพื่อป้องกันมลพิษของ RNase RNA ที่แยกออกจากตัวอย่างที่มี RNase สูง (เช่น ตับอ่อน) จำเป็นต้องมีการจัดเก็บฟอร์มาลดีไฮด์เพื่อเก็บ RNA คุณภาพสูง ซึ่งยิ่งจำเป็นสำหรับการจัดเก็บในระยะยาวโดยพื้นฐานแล้ว RNA ที่สกัดจากตับของหนูจะถูกย่อยสลายหลังจากเก็บไว้ในน้ำหนึ่งสัปดาห์ ในขณะที่ RNA ที่สกัดจากม้ามของหนูยังคงเสถียรหลังจากเก็บไว้ในน้ำเป็นเวลาสามปีนอกจากนี้ การถอดเสียงที่มีขนาดใหญ่กว่า 4kb ยังไวต่อการติดตามการย่อยสลายของ RNase มากกว่าการถอดเสียงที่มีขนาดเล็กเพื่อเพิ่มความเสถียรของตัวอย่าง RNA ที่เก็บ RNA สามารถละลายในเมทาลามีนของไอออน และเก็บไว้ที่ -70 °Cไทไลด์ที่ใช้ในการบันทึก RNA ต้องไม่มีวัตถุเบ็ดเตล็ดที่ย่อยสลาย RNARNA ซึ่งได้มาจากตับอ่อนสามารถเก็บไว้ในเมทาลามีนได้อย่างน้อยหนึ่งปีเมื่อคุณพร้อมที่จะใช้ RNA คุณสามารถใช้วิธีการต่อไปนี้เพื่อทำให้ RNA ตกตะกอน: เติม NaCl เป็น 0.2m และ 4 เท่าของปริมาตรเอทานอล ตั้งอุณหภูมิห้องเป็นเวลา 3-5 นาที และหมุนเหวี่ยง 10,000 × g เป็นเวลา 5 นาที
2. ใช้ reverse transcriptase โดยไม่มีกิจกรรม RNaseH (RNaseH-):
สารยับยั้ง RNase มักถูกเติมลงในปฏิกิริยาการถอดรหัสแบบย้อนกลับเพื่อเพิ่มความยาวและผลผลิตของการสังเคราะห์ cDNAสารยับยั้ง RNase ถูกเพิ่มในปฏิกิริยาการสังเคราะห์แบบลูกโซ่ครั้งแรกโดยมีบัฟเฟอร์และสารรีดิวซ์ เช่น DTT เนื่องจากกระบวนการสังเคราะห์ pre-cDNA ทำลายสารยับยั้ง ดังนั้นจึงปล่อย RNases ที่ถูกผูกไว้ซึ่งย่อยสลาย RNAสารยับยั้งโปรตีน RNase จะป้องกันการสลายตัวของ RNA โดย RNase A, B, C เท่านั้น และไม่ป้องกัน RNases บนผิวหนัง ดังนั้นควรระมัดระวังไม่ให้นำ RNases จากนิ้วแม้ว่าจะใช้สารยับยั้งเหล่านี้ก็ตาม
Reverse transcriptase เร่งปฏิกิริยาการแปลง RNA เป็น cDNAทั้ง M-MLV และ AMV มีกิจกรรม RNaseH ภายนอกนอกเหนือจากกิจกรรมของพอลิเมอเรสของตัวเองกิจกรรม RNaseH แข่งขันกับกิจกรรมของพอลิเมอเรสสำหรับสาย heterozygous ที่เกิดขึ้นระหว่างเทมเพลต RNA และไพรเมอร์ DNA หรือสายต่อ cDNA และลดระดับ RNA: RNA strands ใน DNA complexesเทมเพลต RNA ที่เสื่อมโทรมโดยกิจกรรม RNaseH ไม่สามารถใช้เป็นสารตั้งต้นที่มีประสิทธิภาพสำหรับการสังเคราะห์ cDNA ได้อีกต่อไป ซึ่งจะลดผลผลิตและความยาวของการสังเคราะห์ cDNAดังนั้นการกำจัดหรือลดกิจกรรม RNaseH ของเอนไซม์ย้อนกลับลงอย่างมากจะเป็นประโยชน์อย่างมาก
SuperScriptⅡ reverse transcriptase, MMLV reverse transcriptase ของ RNaseH- และ thermoScript reverse transcriptase, AMV ของ RNaseH- ให้ cDNA แบบเต็มความยาวมากกว่า MMLV และ AMVความไวของ RT-PCR ได้รับผลกระทบจากปริมาณของ cDNA ที่สังเคราะห์ขึ้นThermoScript มีความไวมากกว่า AMVขนาดของผลิตภัณฑ์ RT-PCR ถูกจำกัดโดยความสามารถของรีเวิร์สทรานสคริปเทสในการสังเคราะห์ cDNA โดยเฉพาะอย่างยิ่งเมื่อทำการโคลน Cdnas ที่มีขนาดใหญ่ขึ้นเมื่อเปรียบเทียบกับ MMLV แล้ว SuperScripⅡ เพิ่มผลผลิตของผลิตภัณฑ์ RT-PCR แบบยาวได้อย่างมากรีเวิร์สทรานสคริปเทสของ RNaseH- ยังเพิ่มความคงตัวทางความร้อน ดังนั้นปฏิกิริยาจึงสามารถดำเนินการได้ที่อุณหภูมิสูงกว่าปกติที่ 37-42 ℃ภายใต้เงื่อนไขการสังเคราะห์ที่แนะนำ ไพรเมอร์ oligo(dT) และ 10μCi [alpha-p]dCTP ถูกนำมาใช้การผลิตทั้งหมดของห่วงโซ่แรกคำนวณโดยใช้วิธีการตกตะกอนของ TCAวิเคราะห์ cDNA แบบเต็มความยาวโดยใช้การกำจัดแถบที่เรียงลำดับตามขนาดและการนับจำนวนในเจลอะกาโรสอัลคาไลน์
3. เพิ่มอุณหภูมิการเก็บรักษาความร้อนของการถอดรหัสแบบย้อนกลับ:
อุณหภูมิการกักเก็บที่สูงขึ้นจะช่วยเปิดโครงสร้างทุติยภูมิของ RNA และเพิ่มผลผลิตของปฏิกิริยาสำหรับเทมเพลต RNA ส่วนใหญ่ การถือ RNA และไพรเมอร์ที่อุณหภูมิ 65°C โดยไม่มีบัฟเฟอร์หรือเกลือ แล้วทำให้เย็นลงอย่างรวดเร็วบนน้ำแข็งจะกำจัดโครงสร้างทุติยภูมิส่วนใหญ่และช่วยให้ไพรเมอร์จับตัวกันอย่างไรก็ตาม แม่แบบบางส่วนยังคงมีโครงสร้างรองอยู่ แม้ว่าจะมีการเสียสภาพธรรมชาติเนื่องจากความร้อนแล้วก็ตามการขยายของแม่แบบที่ยากเหล่านี้สามารถทำได้โดยใช้ ThermoScript reverse transcriptase และโดยการวางปฏิกิริยาย้อนกลับของ transcriptase ที่อุณหภูมิสูงขึ้นเพื่อปรับปรุงการขยายสัญญาณอุณหภูมิการกักเก็บที่สูงขึ้นยังสามารถเพิ่มความจำเพาะ โดยเฉพาะอย่างยิ่งเมื่อดำเนินการสังเคราะห์ cDNA โดยใช้ไพรเมอร์เฉพาะยีน (GSPS) (ดูบทที่ 3)หากใช้ GSP ตรวจสอบให้แน่ใจว่าค่า Tm ของไพรเมอร์เท่ากับอุณหภูมิที่คาดไว้ห้ามใช้ oligo(dT) และไพรเมอร์แบบสุ่มที่อุณหภูมิสูงกว่า 60℃ไพรเมอร์แบบสุ่มต้องเก็บไว้ที่ 25°C เป็นเวลา 10 นาทีก่อนที่จะเพิ่มเป็น 60°Cนอกจากการใช้อุณหภูมิการถอดความแบบย้อนกลับที่สูงขึ้นแล้ว ความจำเพาะสามารถปรับปรุงได้โดยการถ่ายโอนส่วนผสม RNA/ ไพรเมอร์โดยตรงจากอุณหภูมิที่ทำให้เสียสภาพ 65°C ไปยังอุณหภูมิที่จับยึดการถอดความแบบย้อนกลับ และเพิ่มส่วนผสมของปฏิกิริยา 2x ที่อุ่นล่วงหน้า (การสังเคราะห์การเริ่มต้นด้วยความร้อน cDNA)วิธีนี้ช่วยป้องกันการจับคู่เบสระหว่างโมเลกุลที่เกิดขึ้นที่อุณหภูมิต่ำกว่าการใช้เครื่องมือ PCR ช่วยลดความซับซ้อนของสวิตช์อุณหภูมิจำนวนมากที่จำเป็นสำหรับ RT-PCR
พอลิเมอเรสที่เสถียรด้วยความร้อน Tth ทำหน้าที่เป็น DNA พอลิเมอเรสเมื่อมี Mg2+ และ RNA พอลิเมอเรสเมื่อมี Mn2+สามารถเก็บความร้อนได้สูงถึง 65 ℃อย่างไรก็ตาม การมีอยู่ของ Mn2+ ในระหว่าง PCR จะลดความเที่ยงตรง ซึ่งทำให้ Tth polymerase ไม่เหมาะสำหรับการขยายสัญญาณที่มีความแม่นยำสูง เช่น การโคลน cDNAนอกจากนี้ Tth ยังมีประสิทธิภาพน้อยกว่าในการถอดรหัสแบบย้อนกลับ ซึ่งลดความไว และเนื่องจากเอนไซม์ตัวเดียวสามารถทำการถอดรหัสแบบย้อนกลับและ PCR ได้ ปฏิกิริยาควบคุมที่ไม่มีการถอดรหัสแบบย้อนกลับจึงไม่สามารถนำมาใช้แยกแยะผลิตภัณฑ์ที่ขยายของ cDNA จากดีเอ็นเอจีโนมที่ปนเปื้อนได้
4. สารเติมแต่งที่ส่งเสริมการถอดความแบบย้อนกลับ:
การเติมสารเติมแต่ง ซึ่งรวมถึงกลีเซอรีนและ DMSO ในปฏิกิริยาการสังเคราะห์ลูกโซ่ครั้งแรกสามารถลดความเสถียรของสายคู่ของกรดนิวคลีอิกและทำให้โครงสร้างทุติยภูมิของ RNA คลายตัวได้สามารถเพิ่มกลีเซอรีนได้ถึง 20% หรือ 10% DMSO โดยไม่ส่งผลกระทบต่อการทำงานของ SuperScriptⅡ หรือ MMLVAMV ยังสามารถทนต่อกลีเซอรอลได้ถึง 20% โดยไม่ลดกิจกรรมเพื่อเพิ่มความไวของ RT-PCR สูงสุดในปฏิกิริยาการถอดความย้อนกลับ SuperScriptⅡ สามารถเติมกลีเซอรอล 10% และหุ้มฉนวนที่ 45 ℃หากเพิ่ม 1/10 ของผลิตภัณฑ์จากปฏิกิริยารีโทรทรานสคริปต์ลงใน PCR ความเข้มข้นของกลีเซอรอลในปฏิกิริยาแอมพลิฟายเออร์คือ 0.4% ซึ่งไม่เพียงพอที่จะยับยั้ง PCR
5. การประมวลผล RNaseH:
ความไวสามารถปรับปรุงได้โดยการรักษาปฏิกิริยาการสังเคราะห์ cDNA ด้วย RNaseH ก่อน PCRสำหรับแม่แบบบางส่วน เชื่อกันว่า RNA ในปฏิกิริยาการสังเคราะห์ cDNA ป้องกันการจับตัวกันของผลิตภัณฑ์ที่ขยาย ซึ่งในกรณีนี้การบำบัดด้วย RNaseH สามารถเพิ่มความไวได้โดยทั่วไป การรักษา RNaseH เป็นสิ่งจำเป็นสำหรับการขยายแม่แบบเป้าหมาย cDNA แบบเต็มความยาวที่ค่อนข้างยาว เช่น tuberous scherosisⅡ ที่มีสำเนาต่ำสำหรับเทมเพลตที่ยากนี้ RNaseH ได้ปรับปรุงสัญญาณที่สร้างโดย cDNA ที่สังเคราะห์โดย SuperScriptⅡ หรือ AMVสำหรับปฏิกิริยา RT-PCR ส่วนใหญ่ การบำบัดด้วย RNaseH เป็นทางเลือก เนื่องจากโดยทั่วไปแล้วขั้นตอนการทำให้เสียสภาพธรรมชาติของ PCR ที่หุ้มฉนวน 95 ℃ จะไฮโดรไลซ์ RNA จาก RNA: DNA complex
6. ปรับปรุงวิธีการตรวจหา RNA จำนวนน้อย:
RT-PCR มีความท้าทายเป็นพิเศษเมื่อมี RNA เพียงเล็กน้อยเท่านั้นการเติมไกลโคเจนเป็นตัวพาในระหว่างการแยก RNA ช่วยเพิ่มผลผลิตของตัวอย่างขนาดเล็กสามารถเพิ่มไกลโคเจนที่ปราศจาก RNase ได้ในเวลาเดียวกันกับ Trizolไกลโคเจนสามารถละลายน้ำได้และสามารถคงอยู่ในเฟสของน้ำได้ด้วย RNA เพื่อช่วยในการตกตะกอนในภายหลังความเข้มข้นที่แนะนำของไกลโคเจนที่ปราศจาก RNase คือ 250μg/ml สำหรับตัวอย่างเนื้อเยื่อน้อยกว่า 50 มก. หรือเซลล์เพาะเลี้ยง 106 เซลล์
การเพิ่ม acetylated BSA เพื่อย้อนกลับปฏิกิริยาการถอดรหัสโดยใช้ SuperScriptⅡ สามารถเพิ่มความไวได้ และสำหรับ RNA จำนวนเล็กน้อย การลดปริมาณของ SuperScriptⅡ และเพิ่ม 40 ยูนิตของ RnaseOut nuclease inhibitor สามารถปรับปรุงระดับการตรวจจับได้หากใช้ไกลโคเจนในการแยก RNA ก็ยังแนะนำให้เพิ่มสารยับยั้ง BSA หรือ RNase เพื่อย้อนกลับปฏิกิริยาการถอดรหัสโดยใช้ SuperScriptⅡ
Ⅱ. เพิ่มความจำเพาะของ RT-PCR
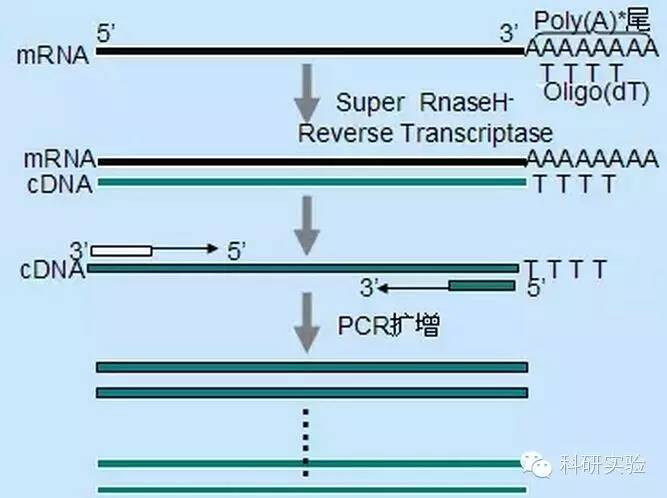
1. การสังเคราะห์ cNDA:
สามารถใช้วิธีการที่แตกต่างกันสามวิธีในการเริ่มต้นการสังเคราะห์ cDNA สายแรก และความจำเพาะสัมพัทธ์ของแต่ละวิธีจะส่งผลต่อปริมาณและประเภทของ cDNA ที่สังเคราะห์ขึ้น
วิธีการไพรเมอร์แบบสุ่มเป็นวิธีที่เฉพาะเจาะจงน้อยที่สุดในสามวิธีไพรเมอร์ถูกอบอ่อนในหลายตำแหน่งตลอดการถอดเสียงเพื่อผลิต cDNA ที่สั้นและมีความยาวบางส่วนวิธีนี้มักใช้เพื่อรับลำดับเทอร์มินัล 5 ′และ cDNA จากเทมเพลต RNA ที่มีขอบเขตโครงสร้างทุติยภูมิหรือกับไซต์สิ้นสุดที่การย้อนกลับของทรานสคริปเทสไม่สามารถทำซ้ำได้เพื่อให้ได้ cDNA ที่ยาวที่สุด อัตราส่วนของไพรเมอร์ต่อ RNA ในตัวอย่าง RNA แต่ละตัวอย่างจำเป็นต้องได้รับการพิจารณาเชิงประจักษ์ความเข้มข้นเริ่มต้นของไพรเมอร์แบบสุ่มมีตั้งแต่ 50 ถึง 250ng ต่อระบบปฏิกิริยา20μlเนื่องจาก cDNA ที่สังเคราะห์จาก RNA ทั้งหมดโดยใช้ไพรเมอร์แบบสุ่มส่วนใหญ่เป็น RNA ไรโบโซม โดยทั่วไปจะเลือกโพลี(A)+RNA เป็นแม่แบบ
การเริ่มต้น Oligo (dT) มีความเฉพาะเจาะจงมากกว่าไพรเมอร์แบบสุ่มมันผสมกับหางโพลี (A) ที่พบที่ปลาย 3 ′ของ mRNA ในเซลล์ยูคาริโอตส่วนใหญ่เนื่องจากโพลี(A)+RNA มีค่าประมาณ 1% ถึง 2% ของ RNA ทั้งหมด ปริมาณและความซับซ้อนของ cDNA จึงน้อยกว่าการใช้ไพรเมอร์แบบสุ่มมากเนื่องจากมีความจำเพาะสูง โดยทั่วไป oligo(dT) จึงไม่ต้องการการปรับให้เหมาะสมสำหรับอัตราส่วน RNA ต่อไพรเมอร์และการเลือกโพลี(A)+ขอแนะนำให้ใช้ 0.5μg oligo(dT) ต่อระบบปฏิกิริยา 20μloligo(dT)12-18 เหมาะสำหรับ RT-PCR ส่วนใหญ่ระบบ ThermoScript RT-PCR ให้ oligo(dT)20 เนื่องจากมีความเสถียรทางความร้อนที่ดีและเหมาะสำหรับอุณหภูมิการถือครองที่สูงขึ้น
ไพรเมอร์เฉพาะยีน (GSP) เป็นไพรเมอร์เฉพาะที่ดีที่สุดสำหรับขั้นตอนการถอดความแบบย้อนกลับGSP เป็นโอลิโกนิวคลีโอไซด์ antisense ที่สามารถไฮบริไดซ์กับลำดับปลายทางของ RNA โดยเฉพาะ แทนที่จะทำให้ Rnas ทั้งหมดหลอมเหลว เช่น ไพรเมอร์แบบสุ่มหรือโอลิโก (dT)กฎที่ใช้ในการออกแบบไพรเมอร์ PCR ยังนำไปใช้กับการออกแบบ GSP ของปฏิกิริยาการถอดรหัสย้อนกลับGSP สามารถเป็นลำดับเดียวกับไพรเมอร์แอมพลิฟายเออร์ที่อบอ่อนที่ส่วนท้ายของ mRNA3 ′หรือ GSP สามารถออกแบบให้อบอ่อนดาวน์สตรีมด้วยไพรเมอร์แอมพลิฟายเออร์ย้อนกลับสำหรับอ็อบเจกต์ขยายบางอย่าง จำเป็นต้องออกแบบไพรเมอร์แอนติเซนส์มากกว่าหนึ่งตัวสำหรับ RT-PCR ที่ประสบความสำเร็จ เนื่องจากโครงสร้างทุติยภูมิของ RNA เป้าหมายอาจป้องกันไม่ให้ไพรเมอร์จับตัวกันขอแนะนำให้ใช้ 1pmol antisense GSP ในระบบปฏิกิริยาการสังเคราะห์ลูกโซ่ตัวแรกที่ 20μl
2. เพิ่มอุณหภูมิการเก็บรักษาความร้อนของการถอดรหัสแบบย้อนกลับ:
เพื่อใช้ประโยชน์จากความจำเพาะของ GSP อย่างเต็มที่ ควรใช้รีเวิร์สทรานสคริปเทสที่มีความเสถียรทางความร้อนสูงรีเวิร์สทรานสคริปเทสที่เสถียรต่อความร้อนสามารถถูกหุ้มฉนวนที่อุณหภูมิสูงขึ้นเพื่อเพิ่มความรุนแรงของปฏิกิริยาตัวอย่างเช่น หาก GSP ถูกอบอ่อนที่อุณหภูมิ 55°C ความจำเพาะของ GSP จะไม่ถูกนำมาใช้อย่างเต็มที่หากดำเนินการถอดความแบบย้อนกลับที่อุณหภูมิ 37°C โดยมีความแม่นยำต่ำโดยใช้ AMV หรือ M-MLVอย่างไรก็ตาม SuperScripⅡ และ ThermoScript สามารถทำปฏิกิริยาที่อุณหภูมิ 50°C หรือสูงกว่า ซึ่งช่วยขจัดผลิตภัณฑ์ที่ไม่เฉพาะเจาะจงที่ผลิตที่อุณหภูมิต่ำกว่าเพื่อความจำเพาะสูงสุด ส่วนผสมของ RNA/ ไพรเมอร์สามารถถ่ายโอนได้โดยตรงจากอุณหภูมิการเสียสภาพธรรมชาติ 65°C ไปยังอุณหภูมิการกักเก็บการถอดความแบบย้อนกลับด้วยการเติมของผสมปฏิกิริยา 2 x ที่อุ่นล่วงหน้า (การเริ่มต้นความร้อนของการสังเคราะห์ cDNA)สิ่งนี้ช่วยป้องกันการจับคู่เบสระหว่างโมเลกุลที่อุณหภูมิต่ำการใช้เครื่องมือ PCR ช่วยลดความยุ่งยากในการเปลี่ยนอุณหภูมิที่จำเป็นสำหรับ RT-PCR
3. ลดการปนเปื้อน DNA ของจีโนม:
ปัญหาที่อาจเกิดขึ้นกับ RT-PCR คือ RNA ปนเปื้อน DNA ของจีโนมการใช้วิธีการแยก RNA ที่ดีกว่า เช่น Trizol Reagent ช่วยลดการปนเปื้อนของ DNA จีโนมในการเตรียม RNAเพื่อหลีกเลี่ยงผลิตภัณฑ์ที่ผลิตจาก DNA จีโนม RNA สามารถบำบัดด้วยแอมพลิฟายเออร์เกรด DnasⅠ เพื่อกำจัด DNA ที่ปนเปื้อนก่อนที่จะทำการถอดความแบบย้อนกลับตัวอย่างถูกเก็บไว้ที่ 65 ℃ใน 2.0mM EDTA เป็นเวลา 10 นาทีเพื่อยุติการย่อย DNaseⅠEDTA คีเลตแมกนีเซียมไอออนเพื่อป้องกันการไฮโดรไลซิสของ RNA ที่ขึ้นอยู่กับแมกนีเซียมไอออนที่เกิดขึ้นที่อุณหภูมิสูง
เพื่อแยก cDNA ที่ขยายแล้วออกจากผลิตภัณฑ์ขยาย DNA ของจีโนม ไพรเมอร์ที่หลอมแยกกับ exon ที่แยกออกมาสามารถออกแบบได้ผลิตภัณฑ์ PCR ที่ได้จาก cDNA จะมีอายุสั้นกว่าผลิตภัณฑ์ที่ได้จาก DNA ของจีโนมที่ปนเปื้อนการทดลองที่มีการควบคุมโดยไม่มีการถอดความแบบย้อนกลับยังดำเนินการกับเทมเพลต RNA แต่ละอันเพื่อพิจารณาว่าชิ้นส่วนที่กำหนดนั้นมาจาก DNA จีโนมหรือ cDNAผลิตภัณฑ์ PCR ที่ได้รับในกรณีที่ไม่มีการถอดความแบบย้อนกลับนั้นได้มาจากจีโนม
สินค้าที่เกี่ยวข้อง
RT-PCR ง่ายᵀᴹฉัน (ขั้นตอนเดียว)
-ชุดอุปกรณ์แบบขั้นตอนเดียวทำให้สามารถดำเนินการถอดความแบบย้อนกลับและ PCR ได้ในหลอดเดียวกันเพียงแค่เพิ่มเทมเพลต RNA, ไพรเมอร์ PCR เฉพาะและ ddH ที่ปราศจาก RNase2O.
- การวิเคราะห์เชิงปริมาณแบบเรียลไทม์ของ RNA สามารถดำเนินการได้อย่างรวดเร็วและแม่นยำ
- ชุดเครื่องมือนี้ใช้รีเอเจนต์การถอดรหัสแบบย้อนกลับของ Foregene และ Foregene HotStar Taq DNA Polymerase รวมกับระบบปฏิกิริยาที่ไม่เหมือนใครเพื่อปรับปรุงประสิทธิภาพการขยายและความจำเพาะของปฏิกิริยาได้อย่างมีประสิทธิภาพ
- ระบบปฏิกิริยาที่ได้รับการปรับปรุงทำให้ปฏิกิริยามีความไวในการตรวจจับสูงขึ้น เสถียรภาพทางความร้อนที่แข็งแกร่งขึ้น และความทนทานที่ดีขึ้น
-ความสามารถในการลบ gDNA อย่างมีประสิทธิภาพ ซึ่งสามารถลบ gDNA ในเทมเพลตได้ภายใน 2 นาที
- ระบบการถอดรหัสแบบย้อนกลับที่มีประสิทธิภาพ ใช้เวลาเพียง 15 นาทีในการสังเคราะห์ cDNA สายแรกให้เสร็จสมบูรณ์
- เทมเพลตที่ซับซ้อน: เทมเพลตที่มีเนื้อหา GC สูงและโครงสร้างรองที่ซับซ้อนสามารถย้อนกลับได้ด้วยประสิทธิภาพสูง
- ระบบการถอดรหัสย้อนกลับความไวสูง เทมเพลตระดับ pg ยังสามารถรับ cDNA คุณภาพสูงได้อีกด้วย
- ระบบการถอดความแบบย้อนกลับมีเสถียรภาพทางความร้อนสูง อุณหภูมิปฏิกิริยาที่เหมาะสมคือ 42 ℃ และยังคงมีประสิทธิภาพการถอดความแบบย้อนกลับที่ดีที่ 50 ℃
เวลาโพสต์: Mar-07-2023