一、เพิ่มความไวของระบบปฏิกิริยา:
1. แยก RNA คุณภาพสูง:
การสังเคราะห์ cDNA ที่ประสบความสำเร็จมาจาก RNA คุณภาพสูงอย่างน้อย RNA คุณภาพสูงควรมีความยาวเต็มและไม่มีตัวยับยั้งการย้อนกลับของทรานสคริปเทส เช่น EDTA หรือ SDSคุณภาพของ RNA จะกำหนดจำนวนสูงสุดของข้อมูลลำดับที่คุณสามารถแปลงเป็น cDNA ได้วิธีการทำให้บริสุทธิ์ RNA ทั่วไปคือวิธีการแบบขั้นตอนเดียวโดยใช้ guanidine isothiocyanate/acid phenolเพื่อป้องกันการปนเปื้อนจากปริมาณ RNase ที่ติดตาม RNA ที่แยกได้จากตัวอย่างที่มี RNase สูง (เช่น ตับอ่อน) จำเป็นต้องเก็บไว้ในฟอร์มาลดีไฮด์เพื่อรักษา RNA คุณภาพสูง โดยเฉพาะอย่างยิ่งสำหรับการจัดเก็บในระยะยาวโดยพื้นฐานแล้ว RNA ที่สกัดจากตับของหนูจะถูกย่อยสลายหลังจากเก็บไว้ในน้ำเป็นเวลาหนึ่งสัปดาห์ ในขณะที่ RNA ที่สกัดจากม้ามของหนูยังคงเสถียรหลังจากเก็บไว้ในน้ำเป็นเวลา 3 ปีนอกจากนี้ การถอดเสียงที่ยาวกว่า 4 kb ยังไวต่อการย่อยสลายโดยการติดตาม RNases มากกว่าการถอดเสียงขนาดเล็กเพื่อเพิ่มความเสถียรของตัวอย่าง RNA ที่เก็บไว้ RNA สามารถละลายใน deionized formamide และเก็บไว้ที่อุณหภูมิ -70°Cฟอร์มาไมด์ที่ใช้ในการเก็บรักษา RNA ต้องปราศจากเศษที่ย่อยสลาย RNARNA จากตับอ่อนสามารถเก็บรักษาไว้ในฟอร์มาไมด์ได้อย่างน้อยหนึ่งปีเมื่อเตรียมใช้ RNA คุณสามารถใช้วิธีต่อไปนี้เพื่อทำให้ RNA ตกตะกอน: เติม NaCl เป็น 0.2M และ 4 เท่าของปริมาตรเอทานอล วางไว้ที่อุณหภูมิห้องเป็นเวลา 3-5 นาที แล้วปั่นแยกที่ 10,000×g เป็นเวลา 5 นาที
2. ใช้ RNaseH-inactive (RNaseH-) reverse transcriptase:
สารยับยั้ง RNase มักถูกเติมลงในปฏิกิริยาการถอดรหัสแบบย้อนกลับเพื่อเพิ่มความยาวและผลผลิตของการสังเคราะห์ cDNAควรเติมสารยับยั้ง RNase ในระหว่างปฏิกิริยาการสังเคราะห์สายแรกโดยมีบัฟเฟอร์และสารรีดิวซ์ (เช่น DTT) เนื่องจากกระบวนการก่อนการสังเคราะห์ cDNA จะทำลายธรรมชาติของสารยับยั้ง ดังนั้นจึงปล่อย RNase ที่ถูกผูกไว้ซึ่งสามารถย่อยสลาย RNA ได้สารยับยั้งโปรตีน RNase ป้องกันการสลายตัวของ RNA โดย RNase A, B, C เท่านั้น และไม่ได้ป้องกัน RNase บนผิวหนัง ดังนั้น ระวังอย่าให้ RNase สัมผัสกับนิ้วของคุณแม้ว่าจะใช้สารยับยั้งเหล่านี้ก็ตาม
Reverse transcriptase กระตุ้นการแปลง RNA เป็น cDNAทั้ง M-MLV และ AMV มีกิจกรรม RNaseH ภายนอกนอกเหนือจากกิจกรรมของพอลิเมอเรสของตัวเองกิจกรรม RNaseH และกิจกรรมโพลีเมอเรสแข่งขันกันเองสำหรับสายไฮบริดที่เกิดขึ้นระหว่างแม่แบบ RNA และ DNA ไพรเมอร์หรือสายขยาย cDNA และลดสาย RNA ใน RNA:DNA คอมเพล็กซ์เทมเพลต RNA ที่เสื่อมโทรมโดยกิจกรรม RNaseH ไม่สามารถทำหน้าที่เป็นสารตั้งต้นที่มีประสิทธิภาพสำหรับการสังเคราะห์ cDNA ได้อีกต่อไป ซึ่งจะลดผลผลิตและความยาวของการสังเคราะห์ cDNAดังนั้นจึงเป็นประโยชน์ในการกำจัดหรือลดกิจกรรม RNaseH ของรีเวิร์สทรานสคริปเทสอย่างมาก。
SuperScript Ⅱ reverse transcriptase, RNaseH- MMLV reverse transcriptase และ thermoScript reverse transcriptase, RNaseH- AMV สามารถรับปริมาณและ cDNA แบบเต็มความยาวได้มากกว่า MMLV และ AMVความไวของ RT-PCR จะได้รับผลกระทบจากปริมาณการสังเคราะห์ cDNAThermoScript มีความไวมากกว่า AMVขนาดของผลิตภัณฑ์ RT-PCR ถูกจำกัดโดยความสามารถของรีเวิร์สทรานสคริปเทสในการสังเคราะห์ cDNA โดยเฉพาะอย่างยิ่งเมื่อทำการโคลน cDNA ที่มีขนาดใหญ่ขึ้นเมื่อเปรียบเทียบกับ MMLV แล้ว SuperScripⅡ เพิ่มผลผลิตของผลิตภัณฑ์ RT-PCR แบบยาวได้อย่างมากนอกจากนี้ RNaseH-reverse transcriptase ยังเพิ่มความสามารถในการทนความร้อน ดังนั้นปฏิกิริยาจึงสามารถทำได้ที่อุณหภูมิสูงกว่าปกติ 37-42°Cภายใต้เงื่อนไขการสังเคราะห์ที่แนะนำ ให้ใช้ไพรเมอร์ oligo(dT) และ 10 μCi ของ [α-P]dCTPผลผลิตรวมของเส้นใยแรกคำนวณโดยใช้วิธีการตกตะกอนแบบ TCAวิเคราะห์ cDNA แบบเต็มความยาวโดยใช้แถบแยกขนาดที่ตัดออกและนับบนเจลอัลคาไลน์อะกาโรส
3. เพิ่มอุณหภูมิการบ่มสำหรับการถอดรหัสแบบย้อนกลับ:
อุณหภูมิการบ่มที่สูงขึ้นจะช่วยเปิดโครงสร้างทุติยภูมิของ RNA ทำให้ผลผลิตของปฏิกิริยาเพิ่มขึ้นสำหรับเทมเพลต RNA ส่วนใหญ่ การบ่ม RNA และไพรเมอร์ที่อุณหภูมิ 65°C โดยไม่มีบัฟเฟอร์หรือเกลือ ตามด้วยการทำให้เย็นลงอย่างรวดเร็วบนน้ำแข็งจะกำจัดโครงสร้างทุติยภูมิส่วนใหญ่และทำให้ไพรเมอร์จับตัวกันอย่างไรก็ตาม แม่แบบบางส่วนยังคงมีโครงสร้างรองอยู่ แม้ว่าจะมีการเสียสภาพธรรมชาติด้วยความร้อนแล้วก็ตามการขยายเทมเพลตที่ยากเหล่านี้สามารถทำได้โดยใช้ ThermoScript Reverse Transcriptase และวางปฏิกิริยาการถอดรหัสย้อนกลับที่อุณหภูมิสูงขึ้นเพื่อปรับปรุงการขยายสัญญาณอุณหภูมิการบ่มที่สูงขึ้นยังสามารถเพิ่มความจำเพาะ โดยเฉพาะอย่างยิ่งเมื่อใช้ไพรเมอร์เฉพาะยีน (GSP) สำหรับการสังเคราะห์ cDNA (ดูบทที่ 3)หากใช้ GSP ตรวจสอบให้แน่ใจว่า Tm ของไพรเมอร์เท่ากับอุณหภูมิการบ่มที่คาดไว้ห้ามใช้ oligo(dT) และไพรเมอร์แบบสุ่มที่อุณหภูมิสูงกว่า 60°Cไพรเมอร์แบบสุ่มต้องการการบ่มที่อุณหภูมิ 25°C เป็นเวลา 10 นาทีก่อนที่จะเพิ่มเป็น 60°Cนอกเหนือจากการใช้อุณหภูมิการถอดความแบบย้อนกลับที่สูงขึ้นแล้ว ความจำเพาะยังสามารถปรับปรุงได้โดยการถ่ายโอนส่วนผสม RNA/ไพรเมอร์โดยตรงจากอุณหภูมิการสลายสภาพธรรมชาติ 65°C ไปยังอุณหภูมิบ่มการถอดรหัสแบบย้อนกลับ และเพิ่มส่วนผสมปฏิกิริยา 2x ที่อุ่นไว้ล่วงหน้า (การสังเคราะห์ cDNA แบบเริ่มร้อน)วิธีนี้ช่วยป้องกันการจับคู่เบสระหว่างโมเลกุลที่เกิดขึ้นที่อุณหภูมิต่ำกว่าการสลับอุณหภูมิหลายรายการที่จำเป็นสำหรับ RT-PCR สามารถทำให้ง่ายขึ้นได้โดยใช้วงจรความร้อน
โพลีเมอเรสทนความร้อน Tth ทำหน้าที่เป็น DNA โพลีเมอเรสเมื่อมี Mg2+ และเป็น RNA โพลีเมอเรสเมื่อมี Mn2+สามารถอุ่นที่อุณหภูมิสูงสุด 65°Cอย่างไรก็ตาม การมีอยู่ของ Mn2+ ในระหว่าง PCR จะลดความเที่ยงตรง ซึ่งทำให้ Tth polymerase ไม่เหมาะสำหรับการขยายสัญญาณที่มีความแม่นยำสูง เช่น การโคลน cDNAนอกจากนี้ Tth ยังมีประสิทธิภาพการถอดรหัสแบบย้อนกลับต่ำ ซึ่งลดความไว และเนื่องจากการถอดรหัสแบบย้อนกลับและ PCR สามารถทำได้ด้วยเอนไซม์ตัวเดียว ปฏิกิริยาควบคุมที่ไม่มีการถอดรหัสแบบย้อนกลับจึงไม่สามารถใช้เพื่อเปรียบเทียบผลิตภัณฑ์ขยาย cDNA กับ DNA จีโนมที่ปนเปื้อนได้ผลิตภัณฑ์ขยายเสียงถูกแยกออกจากกัน
4. สารเติมแต่งที่ส่งเสริมการถอดความแบบย้อนกลับ:
สารเติมแต่งรวมถึงกลีเซอรอลและ DMSO ถูกเติมลงในปฏิกิริยาการสังเคราะห์สายแรก ซึ่งสามารถลดความเสถียรของสายคู่ของกรดนิวคลีอิกและปลดโครงสร้างรองของ RNA ได้สามารถเพิ่มกลีเซอรอลได้สูงสุด 20% หรือ DMSO 10% โดยไม่ส่งผลต่อกิจกรรม SuperScript II หรือ MMLVAMV ยังสามารถทนต่อกลีเซอรอลได้ถึง 20% โดยไม่สูญเสียการทำงานเพื่อเพิ่มความไวของ RT-PCR สูงสุดในปฏิกิริยาการถอดความย้อนกลับของ SuperScriptⅡ สามารถเพิ่มกลีเซอรอล 10% และบ่มที่อุณหภูมิ 45°Cถ้า 1/10 ของผลิตภัณฑ์จากปฏิกิริยาการถอดความย้อนกลับถูกเติมลงใน PCR ความเข้มข้นของกลีเซอรอลในปฏิกิริยาการขยายจะเท่ากับ 0.4% ซึ่งไม่เพียงพอที่จะยับยั้ง PCR
5. RNaseH รักษา:
การรักษาปฏิกิริยาการสังเคราะห์ cDNA ด้วย RNAseH ก่อน PCR สามารถเพิ่มความไวได้สำหรับแม่แบบบางส่วน เชื่อกันว่า RNA ในปฏิกิริยาการสังเคราะห์ cDNA ป้องกันการจับตัวกันของผลิตภัณฑ์ขยาย ซึ่งในกรณีนี้การบำบัดด้วย RNaseH สามารถเพิ่มความไวได้โดยทั่วไป การรักษา RNaseH เป็นสิ่งจำเป็นเมื่อขยายแม่แบบเป้าหมาย cDNA แบบเต็มความยาวที่ยาวขึ้น เช่น low-copy tuberous scherosis IIสำหรับเทมเพลตที่ยากนี้ การรักษา RNaseH ได้ปรับปรุงสัญญาณที่สร้างโดย SuperScript II หรือ cDNA ที่สังเคราะห์ด้วย AMVสำหรับปฏิกิริยา RT-PCR ส่วนใหญ่ การบำบัดด้วย RNaseH เป็นทางเลือก เนื่องจากขั้นตอนการทำให้เสียสภาพธรรมชาติด้วยวิธี PCR ที่อุณหภูมิ 95°C โดยทั่วไปจะไฮโดรไลซ์ RNA ใน RNA:DNA complex
6. การปรับปรุงวิธีการตรวจจับ RNA ขนาดเล็ก:
RT-PCR มีความท้าทายเป็นพิเศษเมื่อมี RNA เพียงเล็กน้อยเท่านั้นไกลโคเจนที่เพิ่มเข้ามาเป็นตัวพาในระหว่างการแยก RNA ช่วยเพิ่มผลผลิตของตัวอย่างขนาดเล็กสามารถเพิ่มไกลโคเจนที่ปราศจาก RNase ได้ในเวลาเดียวกับการเติม Trizolไกลโคเจนสามารถละลายน้ำได้และสามารถเก็บไว้ในเฟสที่เป็นน้ำได้ด้วย RNA เพื่อช่วยในการตกตะกอนในภายหลังสำหรับตัวอย่างเนื้อเยื่อน้อยกว่า 50 มก. หรือเซลล์เพาะเลี้ยง 106 เซลล์ ความเข้มข้นของไกลโคเจนที่ปราศจาก RNase ที่แนะนำคือ 250 ไมโครกรัม/มล.
การเพิ่ม acetylated BSA ในปฏิกิริยาการถอดรหัสย้อนกลับโดยใช้ SuperScript II สามารถเพิ่มความไวได้ และสำหรับ RNA จำนวนเล็กน้อย การลดปริมาณของ SuperScript II และเพิ่ม 40 หน่วยของ RNaseOut nuclease inhibitor สามารถเพิ่มระดับการตรวจจับได้หากใช้ไกลโคเจนในกระบวนการแยก RNA ก็ยังแนะนำให้เพิ่มสารยับยั้ง BSA หรือ RNase เมื่อใช้ SuperScript II สำหรับปฏิกิริยาการถอดรหัสย้อนกลับ
二、เพิ่มความจำเพาะของ RT-PCR
1. การสังเคราะห์ CND:
การสังเคราะห์ cDNA สายแรกสามารถเริ่มต้นได้โดยใช้วิธีการที่แตกต่างกันสามวิธี ซึ่งความจำเพาะสัมพัทธ์ของวิธีการนี้จะส่งผลต่อปริมาณและประเภทของ cDNA ที่สังเคราะห์ขึ้น
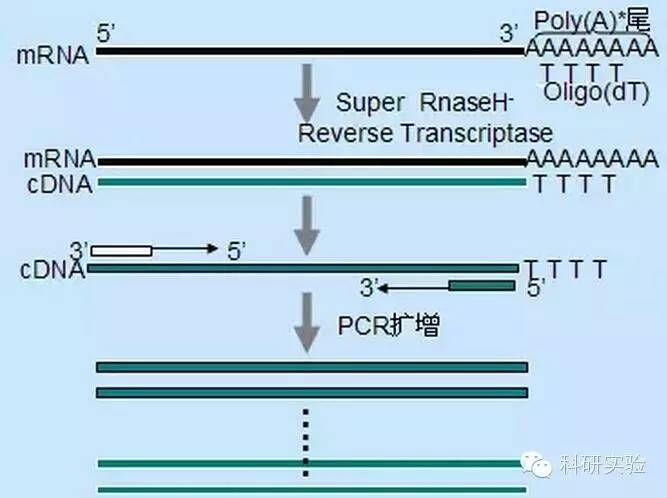
วิธีไพรเมอร์แบบสุ่มมีความเฉพาะเจาะจงน้อยที่สุดในสามวิธีไพรเมอร์หลอมที่ไซต์หลายแห่งตลอดการถอดเสียง สร้าง cDNA ที่สั้นและมีความยาวบางส่วนวิธีนี้มักใช้เพื่อรับลำดับสิ้นสุด 5 'และเพื่อรับ cDNA จากเทมเพลต RNA ที่มีขอบเขตของโครงสร้างรองหรือไซต์สิ้นสุดที่ไม่สามารถจำลองแบบโดย reverse transcriptaseเพื่อให้ได้ cDNA ที่ยาวที่สุด อัตราส่วนของไพรเมอร์ต่อ RNA ในตัวอย่าง RNA แต่ละตัวอย่างจำเป็นต้องได้รับการพิจารณาเชิงประจักษ์ความเข้มข้นเริ่มต้นของไพรเมอร์แบบสุ่มอยู่ในช่วงตั้งแต่ 50 ถึง 250 นาโนกรัมต่อปฏิกิริยา 20 ไมโครลิตรเนื่องจาก cDNA สังเคราะห์จาก RNA ทั้งหมดโดยใช้ไพรเมอร์แบบสุ่มเป็น RNA ของไรโบโซมเป็นหลัก โดยทั่วไปจึงเลือกโพลี(A)+RNA เป็นแม่แบบ
ไพรเมอร์ Oligo(dT) มีความเฉพาะเจาะจงมากกว่าไพรเมอร์แบบสุ่มมันผสมกับหางโพลี (A) ที่พบที่ปลาย 3 ′ของ mRNAs ยูคาริโอตส่วนใหญ่เนื่องจากโพลี(A)+ RNA มีอยู่ประมาณ 1% ถึง 2% ของ RNA ทั้งหมด ปริมาณและความซับซ้อนของ cDNA จึงน้อยกว่าไพรเมอร์แบบสุ่มมากเนื่องจากมีความจำเพาะสูง โดยทั่วไป oligo(dT) จึงไม่ต้องการการปรับอัตราส่วนของ RNA ต่อไพรเมอร์และการเลือก poly(A)+ ให้เหมาะสมขอแนะนำให้ใช้ 0.5μg oligo(dT) ต่อระบบปฏิกิริยา 20μloligo(dT)12-18 เหมาะสำหรับ RT-PCR ส่วนใหญ่ระบบ ThermoScript RT-PCR มี oligo(dT)20 เนื่องจากความเสถียรทางความร้อนที่ดีกว่าสำหรับอุณหภูมิการบ่มที่สูงขึ้น
ไพรเมอร์จำเพาะของยีน (GSP) เป็นไพรเมอร์ที่จำเพาะที่สุดสำหรับขั้นตอนการถอดความแบบย้อนกลับGSP เป็นโอลิโกนิวคลีโอไทด์ antisense ที่สามารถไฮบริไดซ์อย่างเฉพาะเจาะจงกับลำดับเป้าหมาย RNA ซึ่งแตกต่างจากไพรเมอร์แบบสุ่มหรือโอลิโก (dT) ซึ่งจะหลอมรวมกับ RNA ทั้งหมดกฎเดียวกันกับที่ใช้ในการออกแบบไพรเมอร์ PCR ใช้กับการออกแบบ GSP ในปฏิกิริยาการถอดความแบบย้อนกลับGSP สามารถเป็นลำดับเดียวกับไพรเมอร์แอมพลิฟายเออร์ที่หล่อหลอมจนถึงปลายสุด 3′ ของ mRNA หรือสามารถออกแบบ GSP ให้หลอมที่ปลายน้ำของไพรเมอร์แอมพลิฟายเออร์แบบย้อนกลับสำหรับวัตถุที่มีการขยายบางอย่าง จำเป็นต้องออกแบบไพรเมอร์แอนติเซนส์มากกว่าหนึ่งตัวสำหรับ RT-PCR ที่ประสบความสำเร็จ เนื่องจากโครงสร้างทุติยภูมิของ RNA เป้าหมายอาจป้องกันการจับตัวของไพรเมอร์ขอแนะนำให้ใช้ 1 pmol antisense GSP ในปฏิกิริยาการสังเคราะห์สายแรกขนาด 20 ไมโครลิตร
2. เพิ่มอุณหภูมิการบ่มสำหรับการถอดรหัสแบบย้อนกลับ:
เพื่อใช้ประโยชน์อย่างเต็มที่จากประโยชน์เต็มที่ของความจำเพาะของ GSP ควรใช้รีเวิร์สทรานสคริปเทสที่มีความคงตัวทางความร้อนสูงกว่าทรานสคริปเทสย้อนกลับที่ทนความร้อนสามารถบ่มที่อุณหภูมิสูงขึ้นเพื่อเพิ่มความเข้มงวดของปฏิกิริยาตัวอย่างเช่น หาก GSP หลอมที่อุณหภูมิ 55°C ความจำเพาะของ GSP จะไม่ถูกนำมาใช้อย่างเต็มที่หากใช้ AMV หรือ M-MLV สำหรับการถอดความแบบย้อนกลับที่ความเข้มงวดต่ำที่ 37°Cอย่างไรก็ตาม SuperScript II และ ThermoScript สามารถทำปฏิกิริยาได้ที่อุณหภูมิ 50°C หรือสูงกว่า ซึ่งจะกำจัดผลิตภัณฑ์ที่ไม่เฉพาะเจาะจงที่สร้างขึ้นที่อุณหภูมิต่ำกว่าเพื่อความจำเพาะสูงสุด สารผสม RNA/ไพรเมอร์สามารถถ่ายโอนได้โดยตรงจากอุณหภูมิการเสียสภาพธรรมชาติ 65°C ไปยังอุณหภูมิฟักตัวของการถอดความแบบย้อนกลับ และเพิ่มไปยังสารผสมปฏิกิริยา 2x ที่อุ่นไว้ล่วงหน้า (การเริ่มต้นการสังเคราะห์ cDNA แบบร้อน)ซึ่งจะช่วยป้องกันการจับคู่ระหว่างฐานระหว่างโมเลกุลที่อุณหภูมิต่ำการเปลี่ยนอุณหภูมิหลายครั้งที่จำเป็นสำหรับ RT-PCR สามารถทำให้ง่ายขึ้นได้โดยใช้วงจรความร้อน
3. ลดการปนเปื้อน DNA ของจีโนม:
ปัญหาที่อาจเกิดขึ้นกับ RT-PCR คือการปนเปื้อนของ DNA จีโนมใน RNAการใช้วิธีการแยก RNA ที่ดี เช่น Trizol Reagent จะช่วยลดปริมาณของ DNA จีโนมที่ปนเปื้อนการเตรียม RNAเพื่อหลีกเลี่ยงผลิตภัณฑ์ที่ได้จาก DNA จีโนม RNA สามารถบำบัดด้วย DNase I ระดับการขยายเพื่อกำจัด DNA ที่ปนเปื้อนก่อนที่จะทำการถอดรหัสย้อนกลับการย่อย DNase I ถูกยุติโดยการบ่มตัวอย่างใน 2.0 mM EDTA เป็นเวลา 10 นาทีที่ 65°CEDTA สามารถคีเลตแมกนีเซียมไอออน ป้องกันการไฮโดรไลซิสของ RNA ที่ขึ้นอยู่กับแมกนีเซียมที่อุณหภูมิสูง
เพื่อแยก cDNA ที่ขยายออกจากการปนเปื้อนผลิตภัณฑ์ขยาย DNA ของจีโนม ไพรเมอร์สามารถออกแบบให้แต่ละการหลอมเพื่อแยก exonsผลิตภัณฑ์ PCR ที่ได้จาก cDNA จะมีอายุสั้นกว่าผลิตภัณฑ์ที่ได้จาก DNA ของจีโนมที่ปนเปื้อนนอกจากนี้ การทดลองควบคุมที่ไม่มีการถอดความแบบย้อนกลับได้ดำเนินการในแต่ละเทมเพลต RNA เพื่อตรวจสอบว่าชิ้นส่วนที่ได้รับนั้นมาจาก DNA จีโนมหรือ cDNAผลิตภัณฑ์ PCR ที่ได้รับโดยไม่มีการถอดความแบบย้อนกลับนั้นได้มาจากจีโนม
เวลาโพสต์: พฤษภาคม 16-2023








