ภาพรวม
การจำแนกพืชดัดแปลงพันธุกรรมอย่างรวดเร็ว
ข้อความ/ถง หยู่เฉิง
ปฏิบัติการทดลอง/หาน หยิง
บรรณาธิการ / เหวิน ยูจุน
คำ/1600+
เวลาอ่านที่แนะนำ/8-10 นาที
การจำแนกพืชดัดแปลงพันธุกรรมอย่างรวดเร็ว
ในฐานะที่เป็นผู้มาใหม่ในห้องปฏิบัติการ ไม่ใช่งานที่ดีที่จะคัดพืชที่เป็นบวกออกจากกลุ่มพืชที่มีอัตราการเปลี่ยนแปลงต่ำประการแรก ต้องสกัด DNA จากตัวอย่างจำนวนมากทีละตัวอย่าง จากนั้นจึงตรวจหายีนแปลกปลอมด้วย PCRอย่างไรก็ตาม ผลลัพธ์มักจะเป็นช่องว่างและแถบที่มีบางรายการในบางครั้ง แต่ไม่สามารถระบุได้ว่ามีการตรวจจับที่พลาดหรือการตรวจจับที่ผิดพลาด.การเผชิญหน้ากับกระบวนการทดลองและผลลัพธ์เช่นนี้ช่วยอะไรไม่ได้มากหรือ?ไม่ต้องกังวล พี่ชายจะสอนวิธีคัดพืชดัดแปลงพันธุกรรมอย่างง่ายดายและแม่นยำ
ขั้นตอนที่ 1: ออกแบบไพรเมอร์ตรวจจับ
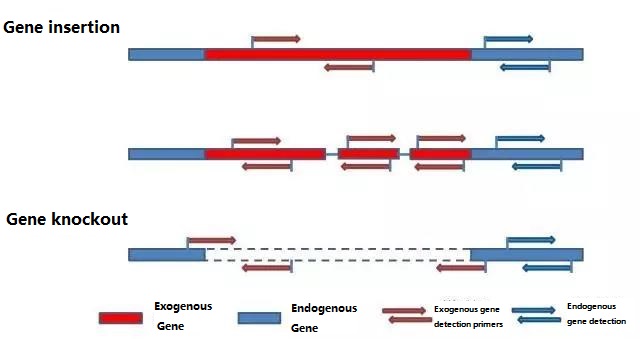
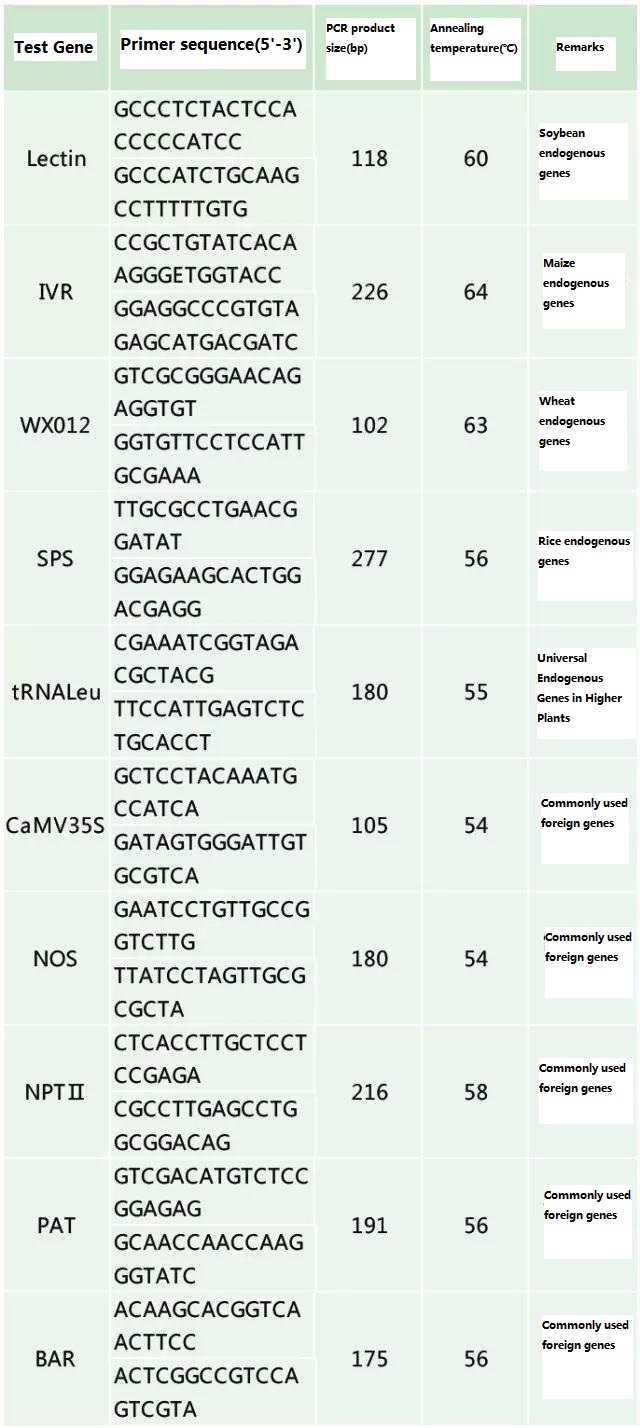
ตรวจสอบยีนภายนอกและยีนภายนอกที่จะตรวจพบตามตัวอย่างที่จะทดสอบ และเลือกลำดับ 100-500bp ที่เป็นตัวแทนในยีนสำหรับการออกแบบไพรเมอร์ไพรเมอร์ที่ดีสามารถรับประกันความถูกต้องของผลการตรวจจับและลดระยะเวลาการตรวจจับ (ดูภาคผนวกสำหรับไพรเมอร์การตรวจจับที่ใช้กันทั่วไป)
บันทึก:
ไพรเมอร์ที่ออกแบบใหม่จำเป็นต้องปรับสภาวะการเกิดปฏิกิริยาให้เหมาะสมและตรวจสอบความถูกต้อง แม่นยำ และขีดจำกัดการตรวจจับของการตรวจจับก่อนที่จะทำการตรวจจับขนาดใหญ่
ขั้นตอนที่ 2:พัฒนาโปรโตคอลการทดลอง

การควบคุมเชิงบวก: ใช้ DNA บริสุทธิ์ที่มีชิ้นส่วนเป้าหมายเป็นแม่แบบเพื่อตรวจสอบว่าระบบปฏิกิริยาและเงื่อนไขของ PCR เป็นปกติหรือไม่
การควบคุมเชิงลบ/ว่าง: ใช้เทมเพลต DNA หรือ ddH2O ที่ไม่มีแฟรกเมนต์เป้าหมายเป็นเทมเพลตเพื่อตรวจสอบว่ามีแหล่งที่มาของการปนเปื้อนในระบบ PCR หรือไม่
การควบคุมอ้างอิงภายใน: ใช้การผสมไพรเมอร์/โพรบของยีนภายนอกของตัวอย่างที่จะทดสอบเพื่อประเมินว่า PCR สามารถตรวจจับแม่แบบได้หรือไม่
บันทึก:
ควรมีการตั้งค่าการควบคุมเชิงบวก ลบ/ว่าง และการควบคุมภายในสำหรับการทดสอบแต่ละครั้งเพื่อประเมินความถูกต้องของผลการทดลอง
ขั้นตอนที่ 3: การเตรียมการทดลอง

ก่อนใช้ให้สังเกตว่าผสมสารละลายอย่างสม่ำเสมอหรือไม่หากพบฝนต้องละลายและผสมตามคำแนะนำก่อนใช้ต้องปิเปตผสม 2×PCR และผสมซ้ำกับไมโครปิเปตก่อนใช้งานเพื่อหลีกเลี่ยงการกระจายไอออนที่ไม่สม่ำเสมอ
บันทึก:
นำคำแนะนำออกมาอ่านอย่างละเอียดและเตรียมการก่อนการทดลองตามคำแนะนำอย่างเคร่งครัด
ขั้นตอนที่ 4: เตรียมระบบปฏิกิริยา PCR
ตามระเบียบการทดลอง ผสมไพรเมอร์ H2O, 2×PCR ผสม, หมุนเหวี่ยงและกระจายไปยังแต่ละหลอดปฏิกิริยา
บันทึก:
สำหรับการทดสอบขนาดใหญ่หรือระยะยาว ขอแนะนำให้ใช้ระบบปฏิกิริยา PCR ที่มีเอนไซม์ UNG ซึ่งสามารถหลีกเลี่ยงการปนเปื้อนละอองที่เกิดจากผลิตภัณฑ์ PCR ได้อย่างมีประสิทธิภาพ
ขั้นตอนที่ 5: เพิ่มเทมเพลตปฏิกิริยา

ด้วยการใช้เทคโนโลยี Direct PCR ไม่จำเป็นต้องมีกระบวนการทำให้บริสุทธิ์ด้วยกรดนิวคลีอิกที่น่าเบื่อสามารถเตรียมแม่แบบตัวอย่างได้ภายใน 10 นาที และเพิ่มลงในระบบปฏิกิริยา PCR ที่สอดคล้องกัน
บันทึก:
วิธีการ Lysis มีผลในการตรวจจับที่ดีกว่า และผลิตภัณฑ์ที่ได้รับสามารถใช้สำหรับปฏิกิริยาการตรวจจับหลายครั้ง
5.1: Direct PCR ของใบ
ตามขนาดรูปภาพในคู่มือ ตัดเนื้อเยื่อใบที่มีเส้นผ่านศูนย์กลาง 2-3 มม. แล้ววางในระบบปฏิกิริยา PCR
หมายเหตุ: ตรวจสอบให้แน่ใจว่าเศษใบไม้แช่อยู่ในสารละลายปฏิกิริยา PCR อย่างสมบูรณ์ และอย่าเพิ่มเนื้อเยื่อใบไม้มากเกินไป
5.2: วิธีการแตกใบ
ตัดเนื้อเยื่อใบที่มีเส้นผ่านศูนย์กลาง 5-7 มม. แล้วใส่ลงในหลอดปั่นแยกหากคุณเลือกใบแก่ โปรดหลีกเลี่ยงการใช้เนื้อเยื่อของเส้นเลือดหลักของใบปิเปต 50ul Buffer P1 ไลเซทในหลอดปั่นแยกเพื่อให้แน่ใจว่าไลเซทสามารถแช่เนื้อเยื่อใบได้อย่างสมบูรณ์ วางไว้ในเครื่องหมุนเวียนความร้อนหรืออ่างโลหะ และไลเซทที่อุณหภูมิ 95°C เป็นเวลา 5-10 นาที


เติมสารละลายที่ทำให้เป็นกลาง 50ul Buffer P2 และผสมให้เข้ากันไลเซทที่เป็นผลลัพธ์สามารถใช้เป็นแม่แบบและเพิ่มลงในระบบปฏิกิริยา PCR
หมายเหตุ: จำนวนแม่แบบควรอยู่ระหว่าง 5-10% ของระบบ PCR และไม่ควรเกิน 20% (ตัวอย่างเช่น ในระบบ PCR 20μl ให้เพิ่ม lysis buffer 1-2μl ไม่เกิน 4μl)
ขั้นตอนที่ 6: ปฏิกิริยา PCR

หลังจากการปั่นแยกหลอดปฏิกิริยา PCR ให้ใส่ในเครื่องมือ PCR เพื่อขยาย
บันทึก:
ปฏิกิริยานี้ใช้แม่แบบที่ไม่ผ่านการทำให้บริสุทธิ์สำหรับการขยาย ดังนั้นจำนวนรอบของการขยายจึงมากกว่า 5-10 รอบกว่าเมื่อใช้แม่แบบ DNA ที่บริสุทธิ์
ขั้นตอนที่ 7: การตรวจจับอิเล็กโทรโฟรีซิสและการวิเคราะห์ผลลัพธ์

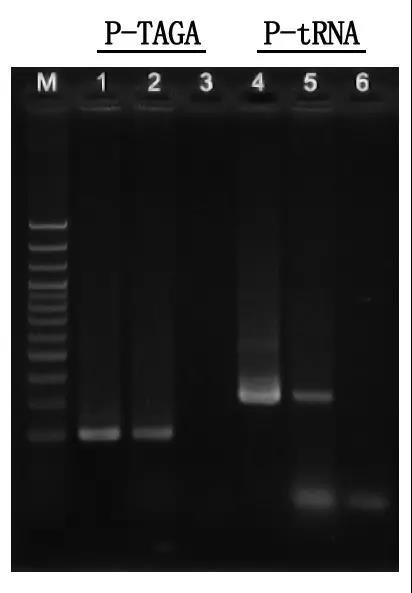
M:100bp บันไดดีเอ็นเอ
1\4: วิธีการทำให้บริสุทธิ์ของ DNA
2\5: วิธี PCR โดยตรง
3\6: การควบคุมว่าง
ควบคุมคุณภาพ:
ผลการทดสอบของชุดควบคุมต่างๆ ในการทดลองควรเป็นไปตามเงื่อนไขต่อไปนี้มิฉะนั้น ควรวิเคราะห์สาเหตุของปัญหา และควรทำการทดสอบอีกครั้งหลังจากกำจัดปัญหาได้แล้ว
ตารางที่ 1 ผลการทดสอบปกติของกลุ่มควบคุมต่างๆ

*เมื่อใช้พลาสมิดเป็นตัวควบคุมเชิงบวก ผลการทดสอบยีนภายนอกอาจเป็นลบ
ผลการตัดสิน:
A. ผลการทดสอบยีนภายนอกของตัวอย่างมีค่าเป็นลบ ซึ่งบ่งชี้ว่าไม่สามารถสกัด DNA ที่เหมาะสมสำหรับการตรวจ PCR ธรรมดาออกจากตัวอย่างได้ หรือ DNA ที่สกัดได้มีสารยับยั้งปฏิกิริยา PCR และควรสกัด DNA อีกครั้ง
B. ผลการทดสอบของยีนภายนอกของตัวอย่างเป็นบวก และผลการทดสอบของยีนภายนอกเป็นลบ ซึ่งบ่งชี้ว่า DNA ที่เหมาะสมสำหรับการตรวจหา PCR ทั่วไปนั้นสกัดมาจากตัวอย่าง และสามารถตัดสินได้ว่าไม่พบยีน XXX ในตัวอย่าง
C. ผลการทดสอบของยีนภายนอกของตัวอย่างเป็นบวก และผลการทดสอบของยีนภายนอกเป็นบวก ซึ่งบ่งชี้ว่า DNA ที่เหมาะสมสำหรับการตรวจหา PCR ทั่วไปได้ถูกแยกออกจากตัวอย่างแล้ว และ DNA ของตัวอย่างมียีน XXXสามารถทำการทดลองยืนยันต่อไปได้
ขั้นตอนที่ 8: ออกแบบไพรเมอร์ตรวจจับ

หลังการทดลองใช้สารละลายโซเดียมไฮโปคลอไรต์ 2% และสารละลายเอทานอล 70% เช็ดบริเวณพื้นที่ทดลองเพื่อป้องกันมลภาวะต่อสิ่งแวดล้อม
ภาคผนวก
ตารางที่ 2 ไพรเมอร์ที่ใช้กันทั่วไปสำหรับการตรวจหา PCR ทั่วไปของพืชดัดแปลงพันธุกรรม
เอกสารอ้างอิง:
SN/T 1202-2010 วิธีการตรวจสอบเชิงคุณภาพ PCR สำหรับส่วนผสมของพืชดัดแปลงพันธุกรรมในอาหาร
ประกาศกระทรวงเกษตรที่ 1485-5-2553 เรื่อง การทดสอบส่วนผสมของพืชดัดแปลงพันธุกรรมและผลิตภัณฑ์ - ข้าว M12 และอนุพันธ์
เวลาโพสต์: Jun-09-2021








