- PCR เป็นวิธีที่ใช้ในการขยาย DNA จากเทมเพลต DNA จำนวนเล็กน้อยRT-PCR ใช้การถอดรหัสแบบย้อนกลับเพื่อสร้างเทมเพลต DNA จากแหล่ง RNA ที่สามารถขยายได้
- โดยทั่วไปแล้ว PCR และ RT-PCR จะเป็นปฏิกิริยาจุดสิ้นสุด ในขณะที่ qPCR และ RT-qPCR ใช้จลนศาสตร์ของอัตราการสังเคราะห์ผลิตภัณฑ์ระหว่างปฏิกิริยา PCR เพื่อหาปริมาณของเทมเพลตที่มีอยู่
- วิธีการใหม่ๆ เช่น PCR ดิจิทัล ให้ปริมาณที่แน่นอนของเทมเพลต DNA เริ่มต้น ในขณะที่วิธีการ เช่น PCR แบบไอโซเทอร์มอลช่วยลดความจำเป็นในการใช้อุปกรณ์ราคาแพงเพื่อให้ได้ผลลัพธ์ที่เชื่อถือได้
ปฏิกิริยาลูกโซ่โพลีเมอเรส (PCR) เป็นเทคนิคอณูชีววิทยาที่ค่อนข้างง่ายและใช้กันอย่างแพร่หลายในการขยายและตรวจจับลำดับ DNA และ RNAเมื่อเปรียบเทียบกับวิธีการโคลนและขยายดีเอ็นเอแบบดั้งเดิม ซึ่งมักใช้เวลาหลายวัน PCR ใช้เวลาเพียงไม่กี่ชั่วโมงPCR มีความไวสูงและต้องใช้เทมเพลตขั้นต่ำในการตรวจจับและขยายลำดับเฉพาะวิธี PCR พื้นฐานมีความก้าวหน้าเพิ่มเติมจากการตรวจจับ DNA และ RNA แบบธรรมดาด้านล่างนี้ เราได้แสดงภาพรวมของวิธี PCR ต่างๆ และรีเอเจนต์ที่เรามอบให้ที่ Enzo Life Sciences สำหรับความต้องการในการวิจัยของคุณเรามุ่งหวังที่จะช่วยให้นักวิทยาศาสตร์เข้าถึงรีเอเจนต์ PCR ได้อย่างรวดเร็วเพื่อใช้ในโครงการวิจัยครั้งต่อไป!
พีซีอาร์
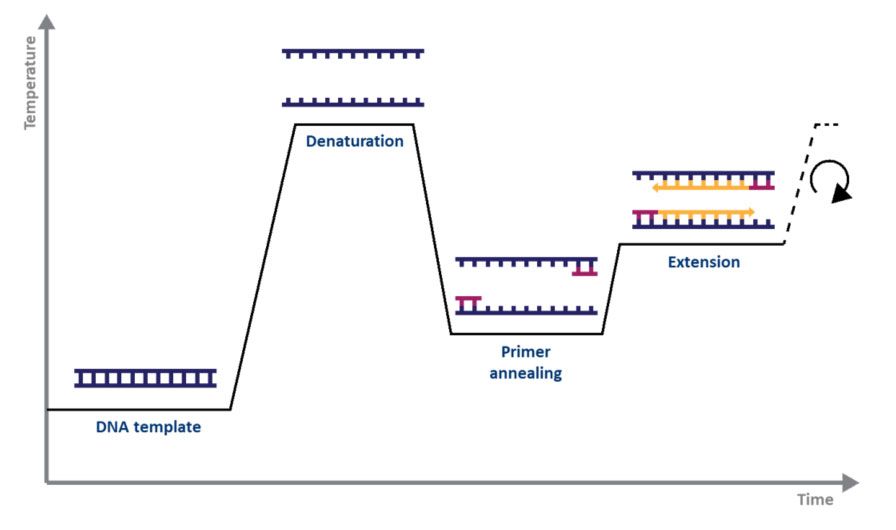
สำหรับ PCR มาตรฐาน สิ่งที่คุณต้องมีคือ DNA polymerase, แมกนีเซียม, นิวคลีโอไทด์, ไพรเมอร์, เทมเพลต DNA ที่จะขยาย และเทอร์โมไซเคิลกลไก PCR นั้นเรียบง่ายตามวัตถุประสงค์: 1) DNA แบบเกลียวคู่ (dsDNA) ถูกทำให้สลายด้วยความร้อน 2) ไพรเมอร์จัดตำแหน่งให้สอดคล้องกับสาย DNA เดี่ยว และ 3) ไพรเมอร์ถูกขยายโดย DNA polymerase ส่งผลให้ได้สำเนาสองชุดของ สายดีเอ็นเอดั้งเดิมกระบวนการสูญเสียสภาพ การหลอม และการยืดตัวในช่วงอุณหภูมิและเวลาต่างๆ เรียกว่าวงจรการขยายสัญญาณหนึ่งรอบ (รูปที่ 1)
| รูปที่ 1.การแสดงวงจรการขยายโดย PCR |
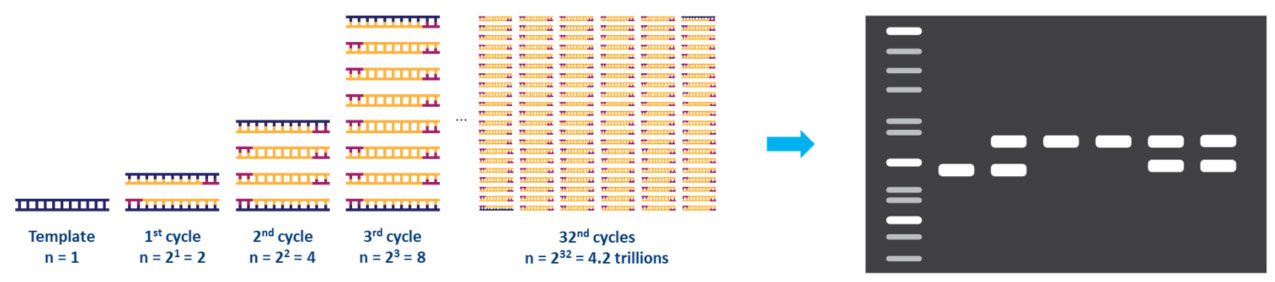
แต่ละขั้นตอนของวงจรควรได้รับการปรับให้เหมาะสมสำหรับเทมเพลตและชุดไพรเมอร์ที่ใช้วงจรนี้จะทำซ้ำประมาณ 20-40 ครั้ง จากนั้นจึงสามารถวิเคราะห์ผลิตภัณฑ์ที่ขยายได้ โดยทั่วไปโดยใช้เจลอะกาโรส (รูปที่ 2)
| รูปที่ 2.การขยายแม่แบบ DNA โดย PCR และการวิเคราะห์โดย agarose gel electrophoresis |
เนื่องจาก PCR เป็นวิธีการที่มีความไวสูงและจำเป็นต้องใช้ปริมาตรที่น้อยมากสำหรับปฏิกิริยาเดี่ยว จึงแนะนำให้เตรียมส่วนผสมหลักสำหรับปฏิกิริยาหลายๆ ปฏิกิริยาส่วนผสมหลักจะต้องผสมให้เข้ากัน จากนั้นจึงแบ่งตามจำนวนปฏิกิริยา เพื่อให้มั่นใจว่าแต่ละปฏิกิริยาจะมีเอนไซม์ dNTP และไพรเมอร์ในปริมาณเท่ากันซัพพลายเออร์หลายราย เช่น Enzo Life Sciences เสนอส่วนผสม PCR ที่มีทุกอย่างอยู่แล้ว ยกเว้นไพรเมอร์และเทมเพลต DNA
บริเวณที่อุดมด้วยกัวนีน/ไซโตซีน (อุดมด้วย GC) แสดงถึงความท้าทายในเทคนิค PCR มาตรฐานลำดับที่อุดมด้วย GC มีความเสถียรมากกว่าลำดับที่มีเนื้อหา GC ต่ำกว่านอกจากนี้ ลำดับที่มี GC มากมีแนวโน้มที่จะสร้างโครงสร้างทุติยภูมิ เช่น ห่วงกิ๊บเป็นผลให้เส้นคู่ที่อุดมด้วย GC นั้นยากต่อการแยกออกอย่างสมบูรณ์ในระหว่างขั้นตอนการทำให้เสียสภาพด้วยเหตุนี้ DNA polymerase จึงไม่สามารถสังเคราะห์สายใหม่ได้โดยไม่มีอุปสรรคอุณหภูมิการเสียสภาพที่สูงขึ้นสามารถปรับปรุงสิ่งนี้ได้ และการปรับอุณหภูมิการหลอมให้สูงขึ้นและระยะเวลาการหลอมที่สั้นลงสามารถป้องกันการจับตัวของไพรเมอร์ที่มี GC มากเป็นพิเศษรีเอเจนต์เพิ่มเติมสามารถเพิ่มประสิทธิภาพการขยายลำดับที่มี GC อยู่มากDMSO กลีเซอรอล และเบทาอีนช่วยทำลายโครงสร้างทุติยภูมิที่เกิดจากปฏิสัมพันธ์ของ GC และด้วยเหตุนี้จึงอำนวยความสะดวกในการแยกเส้นใยสองชั้น
PCR สตาร์ทร้อน
การขยายเสียงที่ไม่เฉพาะเจาะจงเป็นปัญหาที่อาจเกิดขึ้นระหว่าง PCRDNA polymerase ส่วนใหญ่ที่ใช้สำหรับ PCR ทำงานได้ดีที่สุดที่อุณหภูมิประมาณ 68°C ถึง 72°Cอย่างไรก็ตาม เอนไซม์ยังสามารถทำงานได้ที่อุณหภูมิต่ำกว่า แม้ว่าจะต่ำกว่าก็ตามที่อุณหภูมิต่ำกว่าอุณหภูมิการอบอ่อนมาก ไพรเมอร์สามารถจับตัวกันแบบไม่เจาะจงและนำไปสู่การขยายแบบไม่เฉพาะเจาะจง แม้ว่าปฏิกิริยาจะเกิดขึ้นบนน้ำแข็งก็ตามสิ่งนี้สามารถป้องกันได้โดยใช้สารยับยั้งโพลีเมอเรสที่จะแยกตัวออกจาก DNA โพลีเมอเรสเฉพาะเมื่อถึงอุณหภูมิที่กำหนดเท่านั้น จึงเป็นที่มาของคำว่า PCR แบบเริ่มร้อนสารยับยั้งสามารถเป็นแอนติบอดีที่จับโพลีเมอเรสและเปลี่ยนสภาพที่อุณหภูมิการทำให้เสียสภาพเริ่มต้น (โดยทั่วไป 95°C)
โพลีเมอเรสความเที่ยงตรงสูง
แม้ว่า DNA โพลีเมอเรสจะขยายได้ค่อนข้างแม่นยำตามลำดับเทมเพลตดั้งเดิม แต่ข้อผิดพลาดในการจับคู่นิวคลีโอไทด์ก็สามารถเกิดขึ้นได้ความไม่ตรงกันในการใช้งาน เช่น การโคลนอาจส่งผลให้เกิดการถอดเสียงที่ถูกตัดทอน และการแปลผิดหรือโปรตีนที่ไม่ทำงานที่ปลายน้ำเพื่อหลีกเลี่ยงความไม่ตรงกันเหล่านี้ จึงได้มีการระบุโพลีเมอเรสที่มีกิจกรรม "การพิสูจน์อักษร" และรวมไว้ในขั้นตอนการทำงานPfu โพลีเมอเรสสำหรับการพิสูจน์อักษรตัวแรกถูกระบุในปี 1991 ใน Pyrococcus furiosusเอนไซม์ Pfu นี้มีกิจกรรม exonuclease 3 'ถึง 5'เมื่อ DNA ถูกขยายออก เอ็กโซนิวคลีเอสจะกำจัดนิวคลีโอไทด์ที่ไม่ตรงกันที่ปลายสาย 3 ฟุตนิวคลีโอไทด์ที่ถูกต้องจะถูกแทนที่ และการสังเคราะห์ DNA จะดำเนินต่อไปการระบุลำดับนิวคลีโอไทด์ที่ไม่ถูกต้องนั้นขึ้นอยู่กับความสัมพันธ์ในการจับของนิวคลีโอไซด์ไตรฟอสเฟตที่ถูกต้องกับเอนไซม์ ซึ่งการจับที่ไม่มีประสิทธิภาพจะทำให้การสังเคราะห์ช้าลงและทำให้เกิดการทดแทนที่ถูกต้องกิจกรรมการพิสูจน์อักษรของ Pfu polymerase ส่งผลให้เกิดข้อผิดพลาดน้อยลงในลำดับสุดท้ายเมื่อเปรียบเทียบกับ Taq DNA polymeraseในช่วงไม่กี่ปีที่ผ่านมา มีการระบุเอนไซม์พิสูจน์อักษรอื่นๆ และมีการดัดแปลงเอนไซม์ Pfu ดั้งเดิมเพื่อลดอัตราข้อผิดพลาดในระหว่างการขยาย DNA
RT-PCR
Reverse Transcription PCR หรือ RT-PCR อนุญาตให้ใช้ RNA เป็นเทมเพลตได้ขั้นตอนเพิ่มเติมทำให้สามารถตรวจจับและขยาย RNA ได้RNA ถูกคัดลอกแบบย้อนกลับไปยัง DNA เสริม (cDNA) โดยใช้ Reverse transcriptaseคุณภาพและความบริสุทธิ์ของเทมเพลต RNA มีความสำคัญต่อความสำเร็จของ RT-PCRขั้นตอนแรกของ RT-PCR คือการสังเคราะห์ DNA/RNA ลูกผสมReverse transcriptase ยังมีฟังก์ชัน RNase H ซึ่งจะทำให้ส่วน RNA ของลูกผสมลดลงจากนั้นโมเลกุล DNA สายเดี่ยวจะเสร็จสมบูรณ์โดยกิจกรรม DNA polymerase ที่ขึ้นกับ DNA ของ Reverse transcriptase ลงใน cDNAประสิทธิภาพของปฏิกิริยาสายแรกอาจส่งผลต่อกระบวนการขยายสัญญาณจากนี้ไป ขั้นตอน PCR มาตรฐานจะถูกนำมาใช้ในการขยาย cDNAความเป็นไปได้ที่จะเปลี่ยน RNA ให้เป็น cDNA โดย RT-PCR มีข้อดีหลายประการ และส่วนใหญ่จะใช้สำหรับการวิเคราะห์การแสดงออกของยีนRNA เป็นแบบเกลียวเดี่ยวและไม่เสถียรมาก ซึ่งทำให้การทำงานด้วยความท้าทายโดยทั่วไปจะทำหน้าที่เป็นขั้นตอนแรกใน qPCR ซึ่งตรวจวัดปริมาณการถอดเสียง RNA ในตัวอย่างทางชีววิทยา
qPCR และ RT-qPCR
PCR เชิงปริมาณ (qPCR) ใช้ในการตรวจจับ แสดงคุณลักษณะ และหาปริมาณกรดนิวคลีอิกสำหรับการใช้งานหลายประเภทใน RT-qPCR การถอดเสียง RNA มักจะถูกวัดปริมาณโดยการถอดรหัสกลับเข้าไปใน cDNA ก่อน ตามที่อธิบายไว้ข้างต้น จากนั้นจึงดำเนินการ qPCR ในภายหลังเช่นเดียวกับใน PCR มาตรฐาน DNA จะถูกขยายโดยขั้นตอนการทำซ้ำสามขั้นตอน: การเสื่อมสภาพ การหลอม และการยืดตัวอย่างไรก็ตาม ใน qPCR การติดฉลากฟลูออเรสเซนต์ช่วยให้สามารถรวบรวมข้อมูลได้ในขณะที่ PCR ดำเนินไปเทคนิคนี้มีประโยชน์มากมายเนื่องจากมีวิธีการและสารเคมีที่หลากหลาย
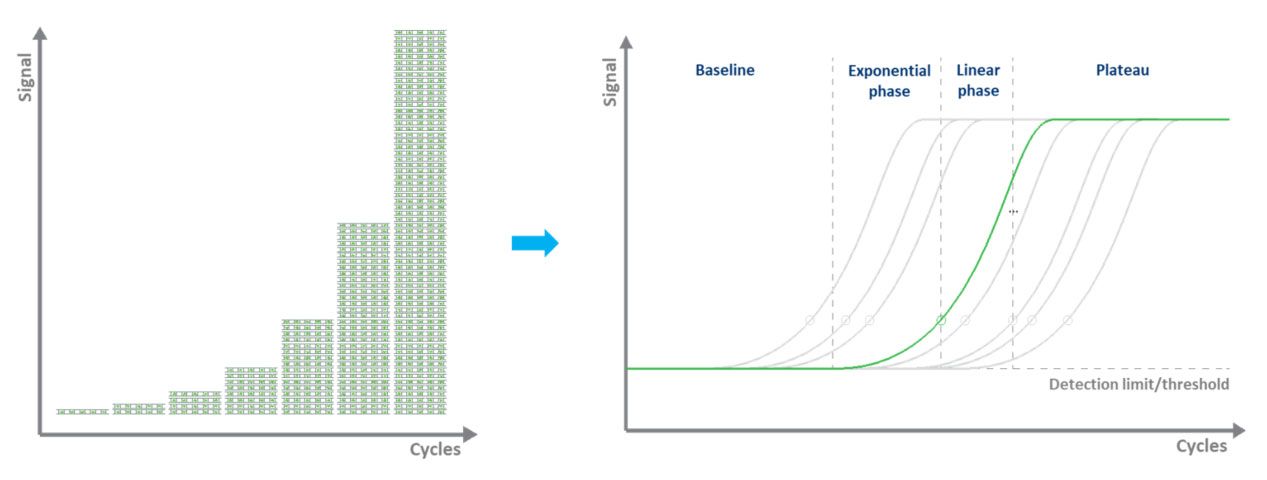
ใน qPCR ที่ใช้สีย้อม (โดยทั่วไปจะเป็นสีเขียว) การติดฉลากเรืองแสงช่วยให้สามารถหาปริมาณของโมเลกุล DNA ที่ถูกขยายได้โดยการใช้สีย้อมจับ dsDNAในแต่ละรอบ จะมีการวัดการเรืองแสงสัญญาณเรืองแสงจะเพิ่มขึ้นตามสัดส่วนของปริมาณ DNA ที่ถูกจำลองดังนั้น DNA จึงถูกวัดปริมาณใน "เรียลไทม์" (รูปที่ 3)ข้อเสียของ qPCR ที่ใช้สีย้อมคือ สามารถตรวจสอบเป้าหมายได้ครั้งละหนึ่งเป้าหมายเท่านั้น และสีย้อมจะจับกับ ds-DNA ใดๆ ที่มีอยู่ในตัวอย่าง
| รูปที่ 3.การขยายเทมเพลต DNA ด้วย qPCR และการวัดสัญญาณเรืองแสงแบบเรียลไทม์ |
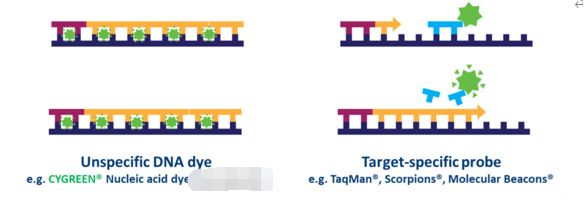
ใน qPCR ที่ใช้โพรบ สามารถตรวจจับเป้าหมายจำนวนมากพร้อมกันในแต่ละตัวอย่าง แต่จำเป็นต้องมีการปรับให้เหมาะสมและการออกแบบโพรบเฉพาะเป้าหมายที่ใช้เพิ่มเติมจากไพรเมอร์มีการออกแบบโพรบหลายประเภท แต่ประเภทที่พบบ่อยที่สุดคือโพรบไฮโดรไลซิส ซึ่งประกอบด้วยฟลูออโรฟอร์และเควนเชอร์การถ่ายโอนพลังงานเรโซแนนซ์เรืองแสง (FRET) ช่วยป้องกันการปล่อยฟลูออโรฟอร์ผ่านตัวดับในขณะที่โพรบยังอยู่ในสภาพสมบูรณ์อย่างไรก็ตาม ในระหว่างปฏิกิริยา PCR โพรบจะถูกไฮโดรไลซ์ในระหว่างการขยายไพรเมอร์และการขยายลำดับเฉพาะที่ยึดกับโพรบความแตกแยกของโพรบจะแยกฟลูออโรฟอร์ออกจากตัวดับ และส่งผลให้การเรืองแสงเพิ่มขึ้นโดยขึ้นอยู่กับการขยาย (รูปที่ 4)ดังนั้น สัญญาณเรืองแสงจากปฏิกิริยา qPCR แบบใช้โพรบจึงเป็นสัดส่วนกับปริมาณของลำดับเป้าหมายของโพรบที่มีอยู่ในตัวอย่างเนื่องจาก qPCR ที่ใช้โพรบมีความเฉพาะเจาะจงมากกว่า qPCR ที่ใช้สีย้อม จึงมักเป็นเทคโนโลยีที่ใช้ในการตรวจวินิจฉัยโดยใช้ qPCR
| รูปที่ 4.ความแตกต่างระหว่าง qPCR แบบใช้สีย้อมและแบบใช้โพรบ |
การขยายไอโซเทอร์มอล
เทคนิค PCR ที่กล่าวถึงข้างต้นต้องใช้อุปกรณ์เทอร์โมไซเคิลราคาแพงเพื่อเพิ่มอุณหภูมิของห้องเพาะเลี้ยงอย่างแม่นยำสำหรับขั้นตอนการทำให้สีอ่อนลง การอบอ่อน และการขยายเทคนิคจำนวนหนึ่งได้รับการพัฒนาซึ่งไม่จำเป็นต้องใช้อุปกรณ์ที่แม่นยำเช่นนั้น และสามารถทำได้ในอ่างน้ำธรรมดาๆ หรือแม้แต่ภายในเซลล์ที่สนใจเทคนิคเหล่านี้เรียกรวมกันว่าการขยายแบบไอโซเทอร์มอล และทำงานโดยอาศัยการขยายแบบเอ็กซ์โปเนนเชียล เชิงเส้น หรือแบบคาสเคด
การขยายเสียงแบบไอโซเทอร์มอลประเภทที่รู้จักกันดีที่สุดคือการขยายแบบไอโซเทอร์มอลแบบใช้ลูปเป็นสื่อกลางหรือ LAMPLAMP ใช้การขยายแบบเอ็กซ์โปเนนเชียลที่อุณหภูมิ 65⁰C เพื่อขยายเทมเพลต DNA หรือ RNAเมื่อดำเนินการ LAMP ไพรเมอร์สี่ถึงหกตัวที่เสริมกับบริเวณของ DNA เป้าหมายจะถูกนำมาใช้ร่วมกับ DNA โพลีเมอเรสเพื่อสังเคราะห์ DNA ใหม่ไพรเมอร์สองตัวนี้มีลำดับอิสระที่จดจำลำดับในไพรเมอร์ตัวอื่นและเชื่อมโยงเข้าด้วยกัน ทำให้เกิดโครงสร้าง "ลูป" ก่อตัวขึ้นใน DNA ที่ถูกสังเคราะห์ขึ้นใหม่ ซึ่งจะช่วยให้ไพรเมอร์หลอมอ่อนในการขยายสัญญาณรอบต่อๆ ไปLAMP สามารถมองเห็นได้หลายวิธี รวมถึงการเรืองแสง เจลอิเล็กโตรโฟรีซิสของอะกาโรส หรือการวัดสีความง่ายในการแสดงภาพและการตรวจจับว่ามีหรือไม่มีผลิตภัณฑ์ด้วยการวัดสีและการขาดแคลนอุปกรณ์ราคาแพง ทำให้ LAMP เป็นตัวเลือกที่เหมาะสมสำหรับการทดสอบ SARS-CoV-2 ในพื้นที่ซึ่งการทดสอบในห้องปฏิบัติการทางคลินิกไม่พร้อม หรือการจัดเก็บและการขนส่งตัวอย่าง ไม่สามารถทำได้หรือในห้องปฏิบัติการที่ก่อนหน้านี้ไม่มีอุปกรณ์เทอร์โมไซคลิก PCR
เวลาโพสต์: 19 ส.ค.-2023