วัสดุเริ่มต้น: RNA
PCR การถอดรหัสย้อนกลับเชิงปริมาณ (RT-qPCR) เป็นวิธีการทดลองที่ใช้ในการทดลอง PCR โดยใช้ RNA เป็นวัสดุเริ่มต้นในวิธีนี้ RNA ทั้งหมดหรือ RNA ของเมสเซนเจอร์ (mRNA) จะถูกแปลงเป็น DNA เสริม (cDNA) ก่อนโดย reverse transcriptaseต่อจากนั้น ปฏิกิริยา qPCR ถูกดำเนินการโดยใช้ cDNA เป็นแม่แบบRT-qPCR ถูกนำมาใช้ในแอปพลิเคชันอณูชีววิทยาที่หลากหลาย รวมถึงการวิเคราะห์การแสดงออกของยีน การตรวจสอบการรบกวน RNA การตรวจสอบความถูกต้องของ microarray การตรวจหาเชื้อโรค การทดสอบทางพันธุกรรม และการวิจัยโรค
วิธีการแบบขั้นตอนเดียวและสองขั้นตอนสำหรับ RT-qPCR
RT-qPCR สามารถทำได้ด้วยวิธีแบบขั้นตอนเดียวหรือสองขั้นตอนขั้นตอนเดียว RT-qPCR รวมการถอดรหัสแบบย้อนกลับและการขยาย PCR ช่วยให้การย้อนกลับของทรานสคริปเทสและ DNA โพลีเมอเรสทำปฏิกิริยาในหลอดเดียวกันภายใต้สภาวะบัฟเฟอร์เดียวกันRT-qPCR ขั้นตอนเดียวต้องใช้ไพรเมอร์เฉพาะลำดับเท่านั้นใน RT-qPCR แบบสองขั้นตอน การถอดความแบบย้อนกลับและการขยาย PCR จะดำเนินการในสองหลอด โดยใช้บัฟเฟอร์ที่เหมาะสมที่สุด สภาวะปฏิกิริยา และกลยุทธ์การออกแบบไพรเมอร์ที่แตกต่างกัน
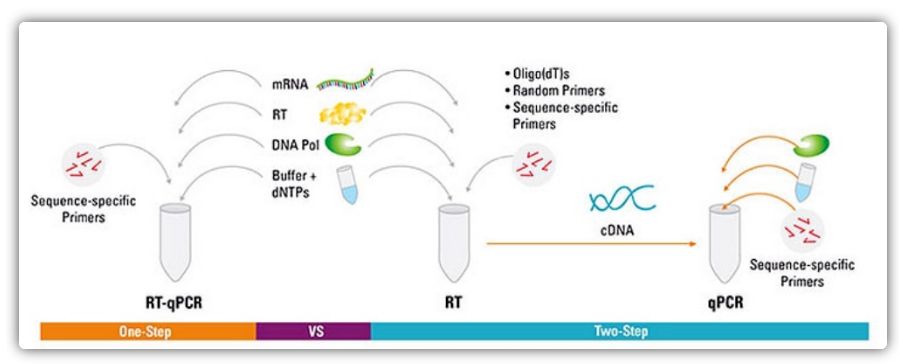
| ข้อได้เปรียบ | ข้อเสีย | |
| ขั้นตอนเดียว | วิธีนี้มีข้อผิดพลาดในการทดลองน้อยกว่าเนื่องจากทั้งสองปฏิกิริยาทำในหลอดเดียว
ขั้นตอนการปิเปตที่น้อยลงช่วยลดความเสี่ยงของการปนเปื้อน
เหมาะสำหรับการขยาย/คัดกรองปริมาณงานสูง รวดเร็วและทำซ้ำได้ | ปฏิกิริยาสองขั้นตอนไม่สามารถเพิ่มประสิทธิภาพแยกกันได้
เนื่องจากสภาวะของปฏิกิริยาถูกทำลายโดยการรวมปฏิกิริยาสองขั้นตอน ความไวจึงไม่ดีเท่าของวิธีการสองขั้นตอน
จำนวนเป้าหมายที่ตรวจพบโดยตัวอย่างเดียวมีน้อย |
| สองขั้นตอน | ความสามารถในการสร้างไลบรารี cDNA ที่เสถียรซึ่งสามารถเก็บไว้ได้เป็นเวลานานและใช้ในหลายๆ ปฏิกิริยา
ยีนเป้าหมายและยีนอ้างอิงสามารถขยายได้จากไลบรารี cDNA เดียวกันโดยไม่จำเป็นต้องใช้ไลบรารี cDNA หลายตัว
บัฟเฟอร์ของปฏิกิริยาและสภาวะของปฏิกิริยาที่เปิดใช้งานการปรับให้เหมาะสมของการทำงานของปฏิกิริยาเดี่ยว
การเลือกเงื่อนไขทริกเกอร์ที่ยืดหยุ่น | การใช้หลอดหลายหลอดและขั้นตอนการปิเปตที่มากขึ้นจะเพิ่มความเสี่ยงของการปนเปื้อนของ DNA และใช้เวลานาน
ต้องการการเพิ่มประสิทธิภาพมากกว่าวิธีการแบบขั้นตอนเดียว |
สินค้าที่เกี่ยวข้อง:
RT-qPCR Easyᵀᴹ (ขั้นตอนเดียว) -SYBR สีเขียว I
RT-qPCR Easyᵀᴹ (ขั้นตอนเดียว) -Taqman
RT Easyᵀᴹ ฉันมาสเตอร์พรีมิกซ์สำหรับการสังเคราะห์ CDNA สายแรก
PCR แบบเรียลไทม์ Easyᵀᴹ-SYBR Green I Kit
PCR แบบเรียลไทม์ Easyᵀᴹ-Taqman
การเลือก RNA และ mRNA ทั้งหมด
เมื่อออกแบบการทดลอง RT-qPCR สิ่งสำคัญคือต้องตัดสินใจว่าจะใช้ RNA ทั้งหมดหรือ mRNA ที่บริสุทธิ์เป็นเทมเพลตสำหรับการถอดความแบบย้อนกลับแม้ว่า mRNA อาจสามารถให้ความไวที่สูงขึ้นเล็กน้อย แต่ RNA ทั้งหมดยังคงใช้อยู่บ่อยครั้งเหตุผลนี้คือ RNA ทั้งหมดมีข้อได้เปรียบที่สำคัญกว่าในฐานะวัสดุเริ่มต้นมากกว่า mRNAประการแรก กระบวนการต้องการขั้นตอนการทำให้บริสุทธิ์น้อยลง ซึ่งช่วยให้มั่นใจได้ถึงการกู้คืนแม่แบบเชิงปริมาณที่ดีขึ้น และการปรับผลลัพธ์ให้เป็นมาตรฐานที่ดีขึ้นสำหรับจำนวนเซลล์เริ่มต้นประการที่สอง หลีกเลี่ยงขั้นตอนการเพิ่มคุณค่า mRNA ซึ่งสามารถหลีกเลี่ยงความเป็นไปได้ของผลลัพธ์ที่เบ้เนื่องจากการคืนค่าที่แตกต่างกันของ mRNA ที่แตกต่างกันโดยรวมแล้ว เนื่องจากในการใช้งานส่วนใหญ่ ปริมาณสัมพัทธ์ของยีนเป้าหมายมีความสำคัญมากกว่าความไวสัมบูรณ์ของการตรวจจับ ดังนั้น RNA ทั้งหมดจึงเหมาะสมกว่าในกรณีส่วนใหญ่
ไพรเมอร์การถอดความแบบย้อนกลับ
ในวิธีการสองขั้นตอน สามารถใช้วิธีการที่แตกต่างกันสามวิธีในการไพรม์ปฏิกิริยา cDNA: ไพรเมอร์โอลิโก (dT) ไพรเมอร์แบบสุ่ม หรือไพรเมอร์เฉพาะลำดับโดยปกติจะใช้ไพรเมอร์ oligo(dT) และไพรเมอร์แบบสุ่มร่วมกันไพรเมอร์เหล่านี้จะหลอมละลายกับแม่แบบ mRNA และจัดเตรียมรีเวิร์สทรานสคริปเทสพร้อมจุดเริ่มต้นสำหรับการสังเคราะห์
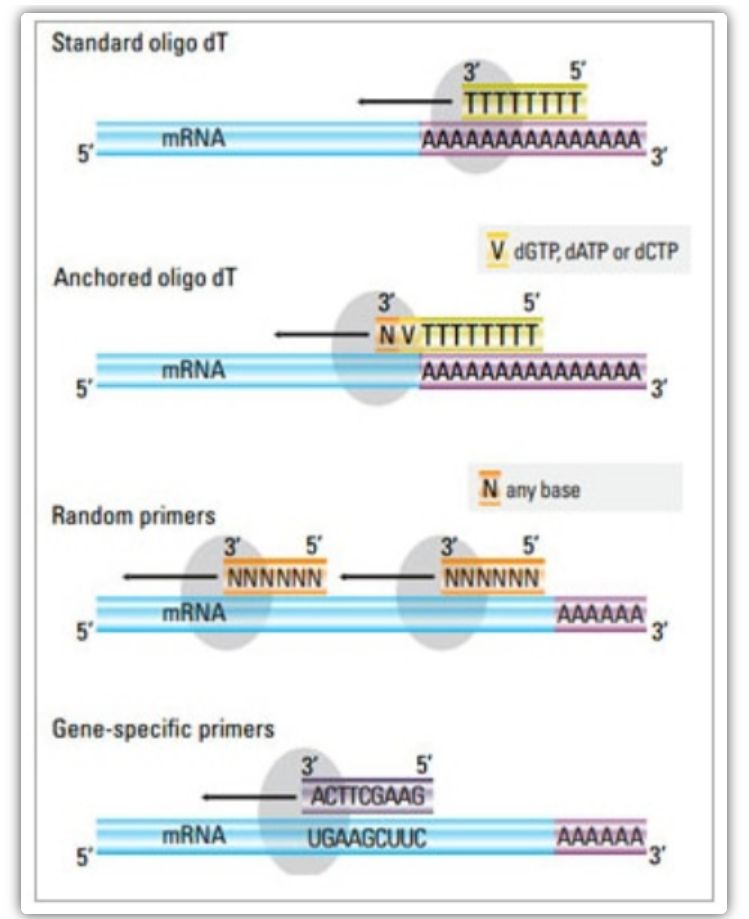
| การเลือกสีรองพื้น | โครงสร้างและหน้าที่ | ข้อได้เปรียบ | ข้อเสีย |
| ไพรเมอร์ Oligo(dT) (หรือไพรเมอร์ oligo(dT) ยึดเกาะ) | การหลอมแบบขยายไปยังไทมีนเรซิดิวที่หางโพลี(A) ของ mRNA;ไพรเมอร์ anchor oligo (dT) มี G, C หรือ A ที่ปลาย 3 ′(จุดยึด) | การสังเคราะห์ cDNA แบบเต็มความยาวจาก mRNA ด้านโพลิ (A)
ใช้ได้เมื่อมีวัสดุเริ่มต้นน้อย
ไซต์ยึดทำให้แน่ใจว่าไพรเมอร์ oligo (dT) ผูกกับหาง 5 'poly (A) ของ mRNA | เหมาะสำหรับการขยายยีนที่มีหางโพลี (A) เท่านั้น
รับ cDNA ที่ถูกตัดทอนจากไซต์รองพื้น*2 ในโพลี(A)
ลำเอียงผูกกับปลาย 3 '*
*ความเป็นไปได้นี้จะลดลงหากใช้ไพรเมอร์แบบแองเคอร์โอลิโก(dT) |
| ไพรเมอร์แบบสุ่ม
| ความยาว 6 ถึง 9 เบส ซึ่งสามารถหลอมไปยังหลาย ๆ ไซต์ระหว่างการถอดรหัส RNA | หลอมรวมเข้ากับ RNA ทั้งหมด (tRNA, rRNA และ mRNA)
เหมาะสำหรับการถอดเสียงที่มีโครงสร้างรองที่สำคัญ หรือเมื่อมีวัสดุเริ่มต้นน้อย
ผลผลิต cDNA สูง | cDNA ถูกคัดลอกย้อนกลับจาก RNA ทั้งหมด ซึ่งโดยปกติไม่ต้องการและอาจทำให้สัญญาณของ mRNA เป้าหมายเจือจางลง
รับ cDNA ที่ถูกตัดทอน |
| ไพรเมอร์เฉพาะลำดับ | ไพรเมอร์แบบกำหนดเองที่กำหนดเป้าหมายลำดับ mRNA เฉพาะ | ไลบรารี cDNA เฉพาะ
ปรับปรุงความไว
การใช้ไพรเมอร์ qPCR แบบย้อนกลับ | จำกัดการสังเคราะห์ยีนเป้าหมายเดียวเท่านั้น |
ทรานสคริปเทสย้อนกลับ
Reverse transcriptase เป็นเอนไซม์ที่ใช้ RNA เพื่อสังเคราะห์ DNAรีเวิร์สทรานสคริปเทสบางตัวมีกิจกรรมของ RNase และสามารถย่อยสลายสาย RNA ในสายไฮบริดของ RNA-DNA หลังจากการถอดรหัสหากไม่มีกิจกรรมของเอนไซม์ RNase ก็สามารถเพิ่ม RNaseH เพื่อประสิทธิภาพ qPCR ที่สูงขึ้นเอ็นไซม์ที่ใช้กันทั่วไป ได้แก่ ไวรัสมะเร็งเม็ดเลือดขาวชนิด Moloney murine รีเวิร์สทรานสคริปเทส และไวรัสเอเวียน มัยอีโลบลาสโตมาสำหรับ RT-qPCR เหมาะอย่างยิ่งที่จะเลือกรีเวิร์สทรานสคริปเทสที่มีความคงตัวทางความร้อนสูงกว่า เพื่อให้การสังเคราะห์ cDNA สามารถทำได้ที่อุณหภูมิสูงขึ้น ทำให้มั่นใจได้ว่าการถอดรหัส RNA ที่ประสบความสำเร็จด้วยโครงสร้างทุติยภูมิที่สูงขึ้น ในขณะที่ยังคงรักษากิจกรรมเต็มที่ตลอดทั้งปฏิกิริยา ส่งผลให้ผลผลิต cDNA สูงขึ้น
สินค้าที่เกี่ยวข้อง:
Foreasy M-MLV Reverse Transcriptase
กิจกรรม RNase H ของเอนไซม์ย้อนกลับ
RNaseH สามารถย่อยสลายสาย RNA จาก RNA-DNA duplexes ทำให้สามารถสังเคราะห์ DNA สายคู่ได้อย่างมีประสิทธิภาพอย่างไรก็ตาม เมื่อใช้ mRNA แบบยาวเป็นเทมเพลต RNA อาจถูกลดประสิทธิภาพก่อนเวลาอันควร ส่งผลให้ cDNA ถูกตัดทอนดังนั้นจึงเป็นประโยชน์ในการลดกิจกรรมของ RNaseH ในระหว่างการโคลน cDNA หากต้องการสังเคราะห์การถอดเสียงแบบยาวในทางตรงกันข้าม รีเวิร์สทรานสคริปเทสที่มีกิจกรรม RNase H มักจะเป็นประโยชน์สำหรับแอปพลิเคชัน qPCR เนื่องจากพวกมันปรับปรุงการหลอมของเพล็กซ์ RNA-DNA ในระหว่างรอบแรกของ PCR
การออกแบบรองพื้น
ไพรเมอร์ PCR ที่ใช้สำหรับขั้นตอน qPCR ใน RT-qPCR ควรได้รับการออกแบบให้ครอบคลุมทางแยก exon-exon ซึ่งไพรเมอร์ขยายสัญญาณอาจขยายขอบเขต exon-intron ที่เกิดขึ้นจริงเนื่องจากไม่มีการขยายลำดับจีโนมของจีโนมที่มีอินตรอน การออกแบบนี้จึงช่วยลดความเสี่ยงของผลบวกลวงที่ขยายจากการปนเปื้อนจีโนมดีเอ็นเอ
ถ้าไพรเมอร์ไม่สามารถออกแบบให้แยก exon หรือ exon-exon ได้ อาจจำเป็นต้องรักษาตัวอย่าง RNA ด้วย DNase-free DNase I หรือ dsDNase เพื่อกำจัดการปนเปื้อนของ DNA จีโนม
การควบคุม RT-qPCR
การควบคุมเชิงลบของการถอดความแบบย้อนกลับ (การควบคุม -RT) ควรรวมอยู่ในการทดลอง RT-qPCR ทั้งหมดเพื่อตรวจหาการปนเปื้อนของ DNA (เช่น จีโนม DNA หรือผลิตภัณฑ์ PCR จากปฏิกิริยาก่อนหน้านี้)การควบคุมนี้มีส่วนประกอบของปฏิกิริยาทั้งหมด ยกเว้นรีเวิร์สทรานสคริปเทสเนื่องจากการถอดความแบบย้อนกลับไม่ได้เกิดขึ้นกับการควบคุมนี้ หากสังเกตเห็นการขยาย PCR จึงมีความเป็นไปได้สูงที่จะมีการปนเปื้อนจาก DNA
เวลาโพสต์: ส.ค.-02-2565








